R statistics/rgrass/es
Esta página se refiere al uso de R dentro de una sesión de GRASS GIS 7 y al uso de GRASS GIS 7 dentro de una sesión de R (vea también R_statistics/spgrass6)
Terminología
El uso de R en conjunto con GRASS GIS puede tener dos significados:
- Usar R dentro de una sesión de GRASS GIS', i.e. inicia R (o RStudio) desde la línea de comandos de GRASS GIS. Puede interesarle esta variante si es primariamente un usuario SIG.
- Usar GRASS GIS dentro de una sesión de R, i.e. se conecta a una localización/directorio de mapas de GRASS GIS desde de R (o RStudio). Puede interesarle esta variante si es priimeramente un usuario de R.
Referencias: ver "Información general" en R_statistics
Instalación
R dentro de GRASS
Usando R dentro de una sesión de GRASS GIS, i.e. se inicia R (o RStudio) desde la línea de comandos de GRASS GIS.
Inicio
- Primero inicie una sesión de GRASS GIS. Luego, en la consola de comandos inicie R (o una sesión de 'rstudio', ver abajo)
- En este ejemplo se usará el conjunto de datos de muestra de North Carolina.
Reiniciar la configuración de la región a la predeterminada
GRASS 7.0.1svn (nc_spm_08_grass7):~ > g.region -d
Lanzar R desde la consola de GRASS
GRASS 7.0.1svn (nc_spm_08_grass7):~ > R
Cargar la librería rgrass7:
> library(rgrass7)
Si quiere seguir el ejemplo usando el conjunto de datos de muestra de North Carolina, cargue la librería rgdal:
> library(rgdal)
Obtener el ambiente de GRASS (directorio de mapas, región, proyección, etc.); puede desplegar los metadatos de su localización mostrando G:
> G <- gmeta()
gisdbase /home/neteler/grassdata location nc_spm_08_grass7 mapset user1 rows 620 columns 1630 north 320000 south 10000 west 120000 east 935000 nsres 500 ewres 500 projection +proj=lcc +lat_1=36.16666666666666 +lat_2=34.33333333333334 +lat_0=33.75 +lon_0=-79 +x_0=609601.22 +y_0=0 +no_defs +a=6378137 +rf=298.257222101 +towgs84=0.000,0.000,0.000 +to_meter=1
Enlistando los mapas existentes
Enlistar los mapas vectoriales existentes:
> execGRASS("g.list", parameters = list(type = "vector"))
Enlistar mapas vectoriales seleccionados (wildcards):
> execGRASS("g.list", parameters = list(type = "vector", pattern = "precip*"))
Guardar mapas vectoriales seleccionados en R:
> my_vmaps <- execGRASS("g.list", parameters = list(type = "vector", pattern = "precip*"))
> attributes(my_vmaps)
> attributes(my_vmaps)$resOut
Enlistar mapas ráster disponibles:
> execGRASS("g.list", parameters = list(type = "raster"))
Enlistar mapas ráster seleccionados (wildcard):
> execGRASS("g.list", parameters = list(type = "raster", pattern = "lsat7_2002*"))
Leyendo datos desde GRASS
Ejemplo 1
Leer en GRASS dos mapas ráster (los mapas "geology_30m" y "elevation" del conjunto de mapas de muestra de North Carolina) en la sesión actual de R como un nuevo objeto "ncdata" (que contiene los dos SpatialGridDataFrames así como los metadatos):
- el parámetro cat indica qué valores ráster se regresarán como factores
# - geology_30m es un mapa categórico (i.e., viene con clases)
# - elevation es una superficie continua
> ncdata <- readRAST(c("geology_30m", "elevation"), cat=c(TRUE, FALSE))
(se puede mostrar una advertencia ya que en el mapa "geology_30m" dos etiquetas no son únicas - como se encuentran en los datos originales.)
Se puede verificar el nuevo objeto R:
> str(ncdata) Formal class 'SpatialGridDataFrame' [package "sp"] with 4 slots ..@ data :'data.frame': 16616 obs. of 2 variables:
y también revisar la estructura de datos en más detalle:
> str(ncdata@data) 'data.frame': 16616 obs. of 2 variables: $ geology_30m: Factor w/ 12 levels "CZfg_217","CZlg_262",..: NA NA NA NA NA NA NA NA NA NA ... $ elevation : num NA NA NA NA NA NA NA NA NA NA ...
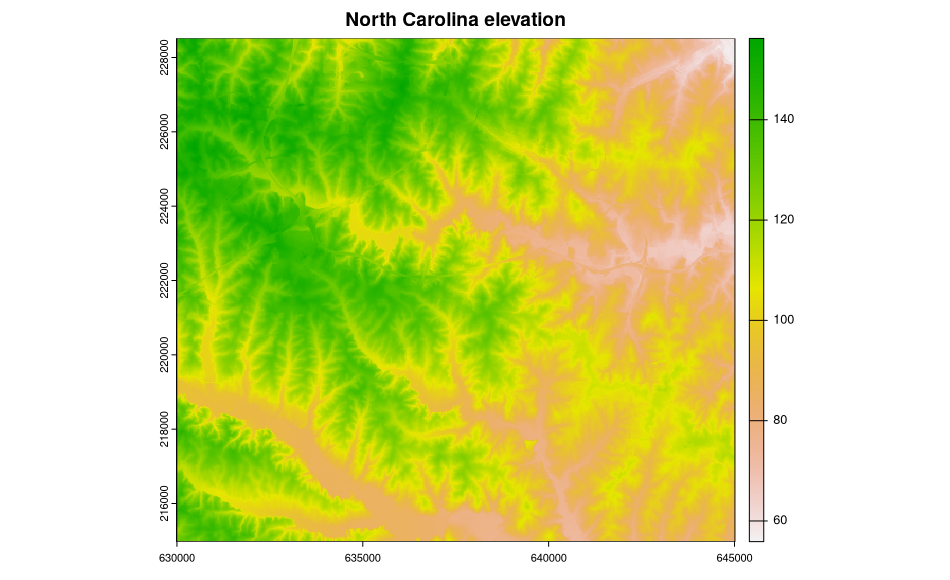
Los metadatos ahora están disponibles, pero no son usados (aún) en para estructurar la clase de objetos sp, e este caso un objeto SpatialGridDataFrame con los datos de dos capas de North Carolina. Aquí la gráfica de los datos de elevación:
> image(ncdata, "elevation", col = terrain.colors(20))
Añadir un título a la gráfica:
> title("North Carolina elevation")
Además, se puede mostrar qué pasa dentro de los objetos leidos en R:
> str(G)
List of 26 $ DEBUG : chr "0" $ LOCATION_NAME: chr "nc_spm_08_grass7" $ GISDBASE : chr "/home/veroandreo/grassdata" $ MAPSET : chr "PERMANENT" $ GUI : chr "wxpython" $ projection : chr "99" $ zone : chr "0" $ n : num 228500 $ s : num 215000 $ w : num 630000 $ e : num 645000 $ t : num 1 $ b : num 0 $ nsres : num 27.5 $ nsres3 : num 10 $ ewres : num 37.5 $ ewres3 : num 10 $ tbres : num 1 $ rows : int 491 $ rows3 : int 1350 $ cols : int 400 $ cols3 : int 1500 $ depths : int 1 $ cells : chr "196400" $ cells3 : chr "2025000" $ proj4 : chr "+proj=lcc +lat_1=36.16666666666666 +lat_2=34.33333333333334 +lat_0=33.75 +lon_0=-79 +x_0=609601.22 +y_0=0 +no_defs +a=6378137 +"| __truncated__ - attr(*, "class")= chr "gmeta"
> summary(ncdata)
Object of class SpatialGridDataFrame
Coordinates:
min max
[1,] 630000 645000
[2,] 215000 228500
Is projected: TRUE
proj4string :
[+proj=lcc +lat_1=36.16666666666666 +lat_2=34.33333333333334
+lat_0=33.75 +lon_0=-79 +x_0=609601.22 +y_0=0 +no_defs +a=6378137
+rf=298.257222101 +towgs84=0.000,0.000,0.000 +to_meter=1]
Grid attributes:
cellcentre.offset cellsize cells.dim
1 630018.8 37.50000 400
2 215013.7 27.49491 491
Data attributes:
geology_30m elevation
CZfg_217:70381 Min. : 55.92
CZig_270:66861 1st Qu.: 94.78
CZbg_405:24561 Median :108.88
CZlg_262:19287 Mean :110.38
CZam_862: 6017 3rd Qu.:126.78
CZbg_910: 4351 Max. :156.25
(Other) : 4942
Ejemplo 2
Aquí hay un ejemplo de un solo mapa transferido desde GRASS GIS a R:
library(rgrass7)
execGRASS("g.region", raster = "elevation", flags = "p")
ncdata <- readRAST("elevation", cat=FALSE)
summary(ncdata)
spplot(ncdata, col = terrain.colors(20))
Resumiendo los datos
Se puede crear una tabla de conteo de celdas:
> table(ncdata$geology_30m)
| CZfg_217 | CZlg_262 | CZig_270 | CZbg_405 | CZve_583 | CZam_720 | CZg_766 | CZam_862 | CZbg_910 | Km_921 | CZbg_945 | CZam_946 | CZam_948 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 70381 | 19287 | 66861 | 24561 | 2089 | 467 | 691 | 6017 | 4351 | 1211 | 1 | 398 | 85 |
Y compararla con el módulo GRASS equivalente:
> execGRASS("r.stats", flags=c("c", "l"), parameters=list(input="geology_30m"), ignore.stderr=TRUE)
217 CZfg 70381 262 CZlg 19287 270 CZig 66861 405 CZbg 24561 583 CZve 2089 720 CZam 467 766 CZg 691 862 CZam 6017 910 CZbg 4351 921 Km 1211 945 CZbg 1 946 CZam 398 948 CZam 85
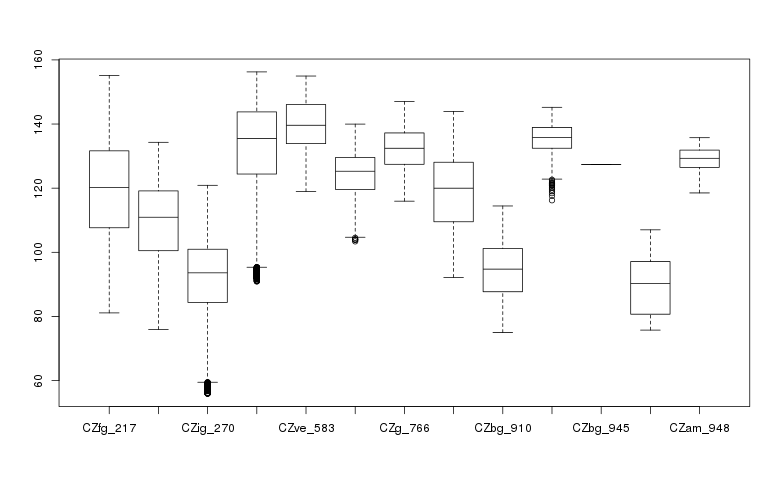
Crear una gráfica de caja y bigotes de los tipos geológicos a diferentes elevaciones:
> boxplot(ncdata$elevation ~ ncdata$geology_30m, medlwd = 1)
Consultando mapas
Algunas veces se quiere consultar mapas de GRASS GIS desde su sesión de R.
Como ejemplo, aquí se transfiere el valor de un pixel ráster a la posición de puntos vectoriales:
# primero definir la región computacional al mapa ráster:
> execGRASS("g.region", raster = "elev_state_500m", flags = "p")
# consulta de mapa ráster en los puntos, transferir los resultados a R
> goutput <- execGRASS("r.what", map="elev_state_500m", points="geodetic_pts", separator=",", intern=TRUE)
> str(goutput)
chr [1:29939] "571530.81289275,265739.968425953,,187.8082200648" ...
# parsearlos
> con <- textConnection(goutput)
> go1 <- read.csv(con, header=FALSE)
> str(go1)
'data.frame': 29939 obs. of 4 variables:
$ V1: num 571531 571359 571976 572391 573011 ...
$ V2: num 265740 265987 267049 267513 269615 ...
$ V3: logi NA NA NA NA NA NA ...
$ V4: Factor w/ 22738 levels "-0.0048115728",..: 6859 6642 6749 6411 6356 6904 7506 7224 6908 7167 ...
> summary(go1)
V1 V2 V3 V4
Min. :121862 Min. : 7991 Mode:logical 0 : 723
1st Qu.:462706 1st Qu.:162508 NA's:29939 * : 293
Median :610328 Median :204502 0.3048 : 47
Mean :588514 Mean :200038 0.6096 : 44
3rd Qu.:717610 3rd Qu.:247633 1.524 : 42
Max. :946743 Max. :327186 0.9144 : 23
(Other):28767
Exportando datos de regreso a GRASS
Finalmente, un objeto SpatialGridDataFrame se escribe de regreso a un mapa ráster GRASS:
Primero preparar algún dato: (raiz cuadrada de la elevación)
> ncdata$sqdem <- sqrt(ncdata$elevation)
Exportar datos desde R de regreso a un mapa ráster GRASS:
> writeRAST(ncdata, "sqdemNC", zcol="sqdem", ignore.stderr=TRUE)
Revisar que la importación a GRASS haya sido correcta:
> execGRASS("r.info", parameters=list(map="sqdemNC"))
+----------------------------------------------------------------------------+ | Map: sqdemNC Date: Sun Jul 19 13:06:34 2015 | | Mapset: PERMANENT Login of Creator: veroandreo | | Location: nc_spm_08_grass7 | | DataBase: /home/veroandreo/grassdata | | Title: ( sqdemNC ) | | Timestamp: none | |----------------------------------------------------------------------------| | | | Type of Map: raster Number of Categories: 0 | | Data Type: DCELL | | Rows: 491 | | Columns: 400 | | Total Cells: 196400 | | Projection: Lambert Conformal Conic | | N: 228500 S: 215000.0002 Res: 27.49490794 | | E: 645000 W: 630000 Res: 37.5 | | Range of data: min = 7.47818253045085 max = 12.5000787351036 | | | | Data Description: | | generated by r.in.bin | | | | Comments: | | | +----------------------------------------------------------------------------+
Usando RStudio en una sesión de GRASS GIS
Si está más acostumbrado a correr R desde RStudio, pero de todas maneras quiere tener todos los datos GRASS disponibles para realizar algún análisis sin perder la posibilidad de usar la línea de comados de GRASS, puede correr:
GRASS> rstudio &
o, si ya está trabajado en algún proyecto de RStudio:
GRASS> rstudio /path/to/project/folder/ &
Luego carque la librería rgrass7 en su proyecto de RStudio
> library(rgrass7)
y listo.
GRASS dentro de R
Usando GRASS GIS dentro de una sesión de R, i.e. se conecta a una localización/directorio de mapas desde adentro de R (o RStudio).
Inicio
En primer lugar, encontrar la ruta a la librería de GRASS GIS. Esta puede ser encontrada facilmente con el siguiente comando (fuera de R, o adentro de R con la llamada system()):
# usuarios de OSGeo4W: nada # usuarios Linux, Mac OSX: grass70 --config path
Debe reportar algo como lo siguiente:
/usr/local/grass-7.0.1
Para llamar la funcionalidad de GRASS GIS 7 desde R, inicie R y use la función initGRASS() para definir la configuración de GRASS:
## usuarios de MS-Windows:
library(rgrass7)
# inicialización y el uso de los datos de muestra de North Carolina
initGRASS(gisBase = "C:/OSGeo4W/apps/grass/grass-7.1.svn",
gisDbase = "C:/Users/marissa/GRASSdata/",
location = "nc_spm_08_grass7", mapset = "user1", SG="elevation")
## usuarios de Linux, Mac OSX:
library(rgrass7)
# inicialización y el uso de los datos de muestra de North Carolina
initGRASS(gisBase = "/usr/local/grass-7.0.1", home = tempdir(),
gisDbase = "/home/veroandreo/grassdata/",
location = "nc_spm_08_grass7", mapset = "user1", SG="elevation")
Nota: el parámetro opcional SG es un objeto 'SpatialGrid' para definir la 'DEFAULT_WIND’ de la localización temporal.
# definir la región computacional a predeterminada (opcional)
system("g.region -dp")
# verificar metadatos
gmeta()
# obtener dos mapas ráster en R
ncdata <- readRAST(c("geology_30m", "elevation"), cat=c(TRUE, FALSE))
# calcular el resumen del objeto
summary(ncdata$geology_30m)
CZfg_217 CZlg_262 CZig_270 CZbg_405 CZve_583 CZam_720 CZg_766 CZam_862
292 78 277 102 8 1 2 25
CZbg_910 Km_921 CZam_946 NA's
18 5 2 1009790
R en GRASS en el modo de bloque (batch)
Corra el siguiente script con
R CMD BATCH batch.R
library(rgrass7)
# inicialización y el uso del cojunto de datos North Carolina
initGRASS(gisBase = "/home/veroandreo/software/grass-7.0.svn/dist.x86_64-unknown-linux-gnu",
home = tempdir(),
gisDbase = "/home/veroandreo/grassdata/",
location = "nc_spm_08_grass7", mapset = "user1", SG="elevation",
override = TRUE)
# definir la región como predeterminada
system("g.region -dp")
# verificar
gmeta()
# leer datos en R
ncdata <- readRAST(c("geology_30m", "elevation"), cat=c(TRUE, FALSE))
# resumen de mapa geológico
summary(ncdata$geology_30m)
proc.time()
El resultado es (no se muestra todo):
cat batch.Rout
R version 3.2.1 (2015-06-18) -- "World-Famous Astronaut"
Copyright (C) 2015 The R Foundation for Statistical Computing
Platform: x86_64-redhat-linux-gnu (64-bit)
...
> library(rgrass7)
Loading required package: sp
Loading required package: XML
GRASS GIS interface loaded with GRASS version: (GRASS not running)
> # initialisation and the use of north carolina dataset
> initGRASS(gisBase = "/home/veroandreo/software/grass-7.0.svn/dist.x86_64-unknown-linux-gnu", home = tempdir(),
+ gisDbase = "/home/veroandreo/grassdata/",
+ location = "nc_spm_08_grass7", mapset = "user1", SG="elevation",
+ override = TRUE)
gisdbase /home/veroandreo/grassdata/
location nc_spm_08_grass7
mapset user1
rows 620
columns 1630
north 320000
south 10000
west 120000
east 935000
nsres 500
ewres 500
projection +proj=lcc +lat_1=36.16666666666666 +lat_2=34.33333333333334
+lat_0=33.75 +lon_0=-79 +x_0=609601.22 +y_0=0 +no_defs +a=6378137
+rf=298.257222101 +towgs84=0.000,0.000,0.000 +to_meter=1
> system("g.region -dp")
projection: 99 (Lambert Conformal Conic)
zone: 0
datum: nad83
ellipsoid: a=6378137 es=0.006694380022900787
north: 320000
south: 10000
west: 120000
east: 935000
nsres: 500
ewres: 500
rows: 620
cols: 1630
cells: 1010600
> gmeta()
gisdbase /home/veroandreo/grassdata/
location nc_spm_08_grass7
mapset user1
rows 620
columns 1630
north 320000
south 10000
...
> ncdata <- readRAST(c("geology_30m", "elevation"), cat=c(TRUE, FALSE))
...
> summary(ncdata$geology_30m)
CZfg_217 CZlg_262 CZig_270 CZbg_405 CZve_583 CZam_720 CZg_766 CZam_862
292 78 277 102 8 1 2 25
CZbg_910 Km_921 CZam_946 NA's
18 5 2 1009790
> proc.time()
user system elapsed
8.061 2.016 10.048
Problemas al correr
Sin espacio en el disco
Linux: un problema común es que /tmp/ sea usado para archivos temporales a pesar de que normalmente es de un pequeño tamaño. Para cambiar a un directorio más grande, puede añadir si directorio $HOME/.bashrc con la entrada:
# cambiar directorio TMO o R (nota: por supuesto que se puede ingresar cualquier otro directorio)
mkdir -p $HOME/tmp
export TMP=$HOME/tmp
La desventaja es que en los sistemas Linux modernos /tmp/ es un disco RAM rápido (basado en tempfs) mientras que los directorios HOME son generalmente más lentos (a menos que tenga una tarjeta SSD). Al menos no se va a terminar el espacio en disco rápidamente.