Temporal data processing/GRASS R raster time series processing/es
GRASS-R / R-GRASS for raster time series processing
Este pequeño ejemplo lo va a guiar en los pasos para exportar un Conjunto de datos Espacio-temporales (strds) guardados en GRASS, importarlos a R, preparar los datos adecuadamente para usar el algoritmo Data INterpolation Empirical Orthogonal Functions (DINEOF) y luego de correrlo, reconstruir la serie de tiempo de rásters, exportarla e importar la nueva strds a GRASS.
Vamos a usar los datos climáticos de North Carolina que están ya disponibles como una Localización GRASS. Los puede obtener de aquí. Si no lo ha hecho, necesita descomprimir el archivo y pegarlo en su carpeta de base de datos d eGRASS (normalmente llamada grassdata).
¿Empezamos?
1. En GRASS GIS
- Abrir GRASS en la Localización/Directorio de mapas de interés
grass70 $HOME/grassdata/nc_climate_spm_2000_2012/climate_1970_2012/
- En listar los mapas ráster con pattern=*tempmean
g.list type=raster pattern=*tempmean
- Crear un strds con la media de los mapas de temperatura: t.create
t.create output=tempmean type=strds temporaltype=absolute \
title="Average temperature" description="Monthly temperature average in NC [deg C]"
- Registrar los mapas en la strds (t.register), enlistar la strds (t.list) y obtener la información general para un conjunto de datos espacio temporales particular (t.info). Más detalles sobre diferentes opciones para registrar mapas en una strds pueden ser encontrados en la wiki particular de map registration.
t.register -i input=tempmean type=raster start=2000-01-01 increment="1 months" \
maps=`g.list type=raster pattern=*tempmean separator=comma`
t.list type=strds
t.info input=tempmean
- Enlistar los mapas y revisar las estadísticas básicas del strds: t.rast.list y t.rast.univar
t.rast.list input=tempmean
t.rast.univar input=tempmean
- Exportar fuera de GRASS: t.rast.export
Como se dijo antes, vamos a usar DINEOF como la técnica para el rellenado de huecos (gap-filling). Este método no está disponible (aún) en GRAS GIS, así que necesitamos exportar la serie de tiempo de rásters'. Para exportar solamente el subconjunto de mapas rásters, definimos la región a la región computacional predeterminada (default) de North Carolina y luego usamos t.rast.export para exportar nuestro strds.
# set default region
g.region -d
t.rast.export input=tempmean output=tempmean4R.tar.gzip compression=gzip
2. Moviendonos a R
- Desde la consola de GRASS, abra R (o rstudio)
Note que puede abrir directamente un proyecto en el que esté trabajando (como en el ejemplo de arriba) o solo escribir R o rstudio seguido de & así no pierde el prompt en la consola de GRASS. Para más detalles y ejemplos de cómo usar R o rstudio dentro de una sesión de grass, puede ver la wiki del tema. Vea las secciones R within GRASS y Rstudio within GRASS.
rstudio $HOME/Documents/foss4g_bonn/ts_grass_r &
Una vez dentro de R, primero vamos a cargar el paquete rgrass7. Esta librería provee la interfaz con GRASS GIS 7. Vamos a revisar alguna información básica de la sesión.
# Cargar el paquete rgrass7
library(rgrass7)
# Revisar el ambiente GRASS
genv <- gmeta()
genv
gisdbase /home/veroandreo/grassdata
location nc_climate_spm_2000_2012
mapset climate_1970_2012
rows 104
columns 139
north 260500
south 208500
west 624500
east 694000
nsres 500
ewres 500
projection +proj=lcc +lat_1=36.16666666666666 +lat_2=34.33333333333334 +lat_0=33.75
+lon_0=-79 +x_0=609601.22 +y_0=0 +no_defs +a=6378137 +rf=298.257222101
+towgs84=0.000,0.000,0.000 +to_meter=1Esto nos da información acerca de la proyección y la región, entre otras cosas que vamos a necesitar más adelate (ver abajo).
Dado que ni los módulos temporales de GRASS, ni los conjuntos de datos temporales de GRASS están implementados (aún) en R, cuando necesite hacer un proceso de una serie de tiempo que tenga en la GRASS DB, debe exportar todos los mapas y luego leerlos en R. Por lo tanto, necesitamos cargar el siguiente paquete también:
# Load other required packages
library(spacetime)
library(raster)
library(rgdal)Ahora, necesitamos leer el strds exportado desde GRASS en R con la función read.tgrass del paquete spacetime.
# Importar strds a R
tempmean_in_R <- read.tgrass("tempmean4R.tar.gzip", localName = FALSE, useTempDir = FALSE)Note que R importa nuestro strds como un RasterStack. Veamos si todo está ahi:
# Obtener información sobre el RasterStack
class(tempmean_in_R)
[1] "RasterStack"
attr(,"package")
[1] "raster"
# Dimensiones
> dim(tempmean_in_R)
[1] 104 139 156Podemos también pedir el resumen de capas particulares en el stack o de todo el RasterStack:
# Resumen de la primer capa
summary(tempmean_in_R[[1]])
X2000_01_tempmean
Min. 2.833784
1st Qu. 3.667942
Median 3.936594
3rd Qu. 4.132093
Max. 4.496206
NA's 0.000000
# Resumen del RasterStack
summary(tempmean_in_R)Y, por supuesto, podemos graficar alguna capa:
# Graficar la 3er capa del stack
plot(tempmean_in_R,3)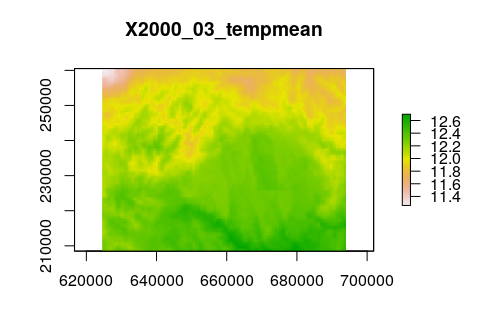
Para la mayoría de los métodos en R (por ej: Theil-Sen slope, fft, dineof) necesitamos transformar nuestro RasterStack en una matriz o data.frame En el caso particular de este ejemplo, necesitamos transformar nuestro RasterStack en una matriz mtx con m = mapas y t = tiempo, i.e.: en esta matriz tenemos nuestros mapas como filas y el tiempo como columnas. Por lo tanto, en cada columna tenemos una serie de tiempo para un pixel dado. Vamos a empezar:
# RasterStack a matriz (esto nos da mapas como columnas y tiempo como filas)
txm_tempmean <- as.matrix(tempmean_in_R)
dim(txm_tempmean)
[1] 14456 156
# Transponemos la matriz (necesitamos tiempo como columnas y mapas como filas)
mxt_tempmean <- t(txm_tempmean)
dim(mxt_tempmean)
[1] 156 14456El punto para DINEOF es rellenar los huecos (gap-filling). Sin embargo, como uestro conjunto de datos proviene de interpolaciones de estaciones climáticas, no tenemos tales huecos...
# Revisar huecos en los datos
sum(is.na(mxt_tempmean))
[1] 0Así que para propósitos de este ejemplo, vamos a crearlos
# Crear huecos
# Conteo total de pixeles: 14456*156=2255136
# Valores NULL deben aparecer como NaN
set.seed(46)
n = 400000
mxt_tempmean[mysamples <- sample(length(mxt_tempmean), n)] <- NaN
sum(is.na(mxt_tempmean))
[1] 400000Ahora estamos listos para usar DINEOF para rellenar los datos faltantes. Puede encontrar más información sobre DINEOF en el sitio oficial. Sin embargo, el paquete de R que provee DINEOF se llama sinkr y puede ser instalado desde aquí.
# Cargar la librería que contiene la fución dineof y otras librerías requeridas
library(sinkr)
library(irlba)
library(Matrix)
# o, descargar la función dineof desde el repositorio y ''source it'':
# source('dineof.r')
# Correr el algoritmo - configuración predeterminada
result_tempmean <- dineof(mxt_tempmean)El objeto result_tempmean es una lista con todos los resultados de dineof. Puede investigarla ud. mismo, pruebe con otra configuración y compare los resultados.
# Explorar qué tiene dentro
names(result_tempmean)
[1] "Xa" "n.eof" "RMS" "NEOF"Para este ejemplo, vamos a extraer solamente la matriz espacio-temporal reconstruida y mostrarla:
# Extraer la matriz rellenada (gap-filled)
tempmean_new <- result_tempmean$Xa
# Definir la paleta de colores
library(RColorBrewer)
pal <- colorRampPalette(c("blue", "cyan", "yellow", "red"))
# Mostrar las matrices espacio-temporales con huecos y sin huecos
par(mfrow = c(2,1))
image(mxt_tempmean, col = pal(100), main = "Original data with gaps")
image(tempmean_new, col = pal(100), main = "Gap-filled data")
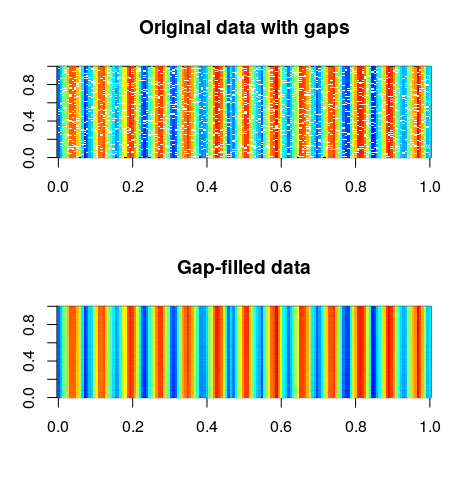
Y queremos ver cómo la serie de tiempo de un pixel particular es reconstruida. Por lo tanto, necesitamos graficar todas las filas de una cierta columna. ¿Recuerdas cómo se formó nuestra matriz?
# Graficar la serie de tiempo de un pixel particular de ambas matrices
pixel <- 10000
ylim <- range(c(mxt_tempmean[,pixel], tempmean_new[,pixel]), na.rm = TRUE)
plot(mxt_tempmean[,pixel], t = "l", ylim = ylim, lwd = 2, ylab = "Temperature", xlab = "Time")
lines(tempmean_new[,pixel], col = 2, lty = 3)
legend(123, 13, c("original","gap-filled"), lty = c(1,3), lwd = c(2,1), col = c(1,2), cex = 0.8)
abline(h = 0, v = 0, col = "gray60")
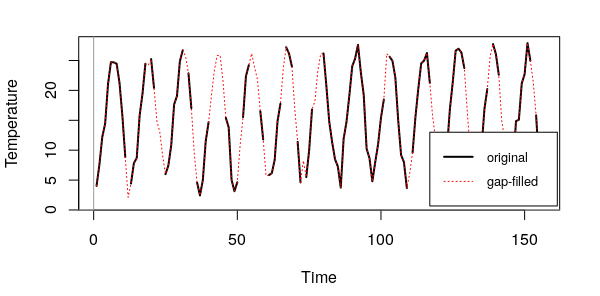
Asumamos que estamos contentos con el resultado, necesitamos regresar a un formato strds. Vamos a reconstruir la serie de tiempo de rásters empezando con una matriz mxt. Para eso, primero creamos rásters vacíos de las mismas dimensiones que nuestros mapas ráster originales y luego los rellenamos con cada fila de la matriz rellenada'. Lo que obtenemos es entonces una lista de rásters. Veamos:
# Crear objetos rásters para rellenar con los datos de tempmean_new
tempmean_2_grass <- raster(nrows = 104, ncols = 139,
xmn = 624500, xmx = 694000,
ymn = 208500, ymx = 260500,
crs = tempmean_in_R)
dim(tempmean_2_grass)
[1] 104 139 1
# Extraer los datos de cada fila de ''tempmean_new'' y construir una capa ráster
# Salida: lista de ráters
tempmean_new_rl <- lapply(1:nrow(tempmean_new), function(i) {
setValues(tempmean_2_grass, tempmean_new[i,])
} )
length(tempmean_new_rl)
[1] 156
dim(tempmean_new_rl[[3]])
[1] 104 139 1
# Graficar una capa ráster de la lista
plot(tempmean_new_rl[[3]], main="X2000_03_tempmean_new")
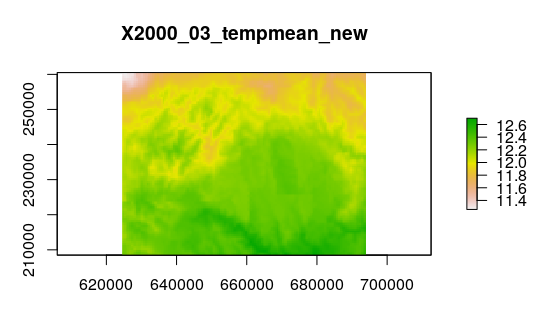
Puede comparar con la tercer capa de los datos originales.
plot(tempmean_in_R,3)Bien, hasta ahora, tenemos una lista de rásters. Pero para exportarlos e importarlos a GRASS, necesitamos de nuevo un RasterStack.
# reconstruir el RasterStack
tempmean_new_rs <- stack(tempmean_new_rl)No solo un RasterStack, pero necesitamos especificar la dimensión temporal, i.e.: necesitamos añadir tiempo al RasterStack'.
time_4_new_rs <- seq(as.Date("2001-01-01"), as.Date("2012-12-01"))
tempmean_new_rs_and_time <- setZ(tempmean_new_rs, time_4_new_rs, name="time")Finalmente, exportamos el RasterStack a GRASS con la función write.tgrass del paquete spacetime.
write.tgrass(tempmean_new_rs_and_time, "tempmean_new_from_R.tar.gzip")3. De regreso a GRASS
Ahora cambiamos de nuevo a la consola de GRASS e importamos el strds con t.rast.import. Este comando creará el strds y registrará todos los mapas que hay en él.
t.rast.import input=tempmean_new_from_R.tar.gz output=tempmean_dineof base=tempmean_dineof extrdir=/tmp
¡Diviertanse!