Processing lidar and UAV point clouds in GRASS GIS (workshop at FOSS4G Boston 2017)/es

Descripción: GRASS GIS ofrece, entre otras cosas una gran cantidad de herramientas analíticas para nubes de puntos, análisis de terreno y percepción remota. En este taller vamos a explorar las herramientas de GRASS GIS para procesar nubes de puntos obtenidas por lidar o procesando imágenes UAV. Vamos a empezar con una breve introducción a la interfaz gráfica (GUI) y continuaremos con una pequeña introducción a la interfaz del intérprete Python de GRASS GIS. Los participantes decidirán si usan la GUI, la línea de comandos, Python o un Jupyter Notebook para el resto del taller. Vamos a explorar las propiedades de la nube de puntos, interpolar superficies y realizar análisis de terreno avanzados para detectar artefactos y formas de terreno (landforms). Vamos a ver diferentes técnicas de visualización 2D y 3D para obtener más información de los datos y a finalizar con análisis de vegetación.
Requisitos: Este taller es accesible a principiantes, pero se requiere algún conocimiento básico de procesamiento de lidar o SIG para que la experiencia sea más provechosa.
Autores: Vaclav Petras, Anna Petrasova, y Helena Mitasova de la North Carolina State University
Contribuyentes: Robert S. Dzur y Doug Newcomb
Preparaciónn
Software
GRASS GIS 7.2 compilado con libLAS (ej. debe funcionar r.in.lidar).
OSGeo-Live
Todo el software está incluido en OSGeo-Live.
Ubuntu
Instalar GRASS GIS desde los paquetes:
sudo add-apt-repository ppa:ubuntugis/ubuntugis-unstable sudo apt-get update sudo apt-get install grass
Linux
Para otras distribuciones de Linux, por favor intente encontrando GRASS GIS en su administrador de paquetes.
MS Windows
Descargue los binarios de GRASS GIS desde grass.osgeo.org.
Mac OS
Instale GRASS GIS usando Homebrew osgeo4mac:
brew tap osgeo/osgeo4mac brew install numpy brew install liblas --build-from-source brew install grass7 --with-liblas
Si ya los tiene instalado, debe usar reinstall en vez de install.
Note que en algunas versiones de Mac OS el módulo r.in.lidar no es accesable, así que tiene que revisar esto y usar r.in.ascii en combinación con las herramientas de la línea de comandos libLAS y PDAL para lograr lo mismo, o de manera preferente, use OSGeo-Live.
Note que actualmente no hay ninguna versión reciente para Mac OS donde sirva la vista 3D.e
Extensiones (Addons)
Debe instalar las siguientes extensiones de GRASS GIS. Esto lo puede hacer a través de la GUI, pero por simplicidad copie, pegue y ejecute las siguientes líneas de comando una por una en la línea de comandos:
g.extension r.geomorphon g.extension r.skyview g.extension r.local.relief g.extension r.shaded.pca g.extension r.area g.extension r.terrain.texture g.extension r.fill.gaps
Para tareas extra:
g.extension v.lidar.mcc
Datos
Va a necesitar los siguientes archivos ZIP, descarguelos y extraigalos:
Para la tarea extra, descargue también este archivo (mucho más grande):
Basic introduction to graphical user interface
GRASS GIS Spatial Database
Aquí vamos a dar una breve introducción a GRASS GIS. Para este ejercicio no es necesario tener una comprensión completa de cómo usar GRASS GIS. Sin embargo, va a necesitar saber colocar sus datos correctamente en un directorio de bases de datos de GRASS GIS, así como algunas funcionalidades básicas de GRASS.
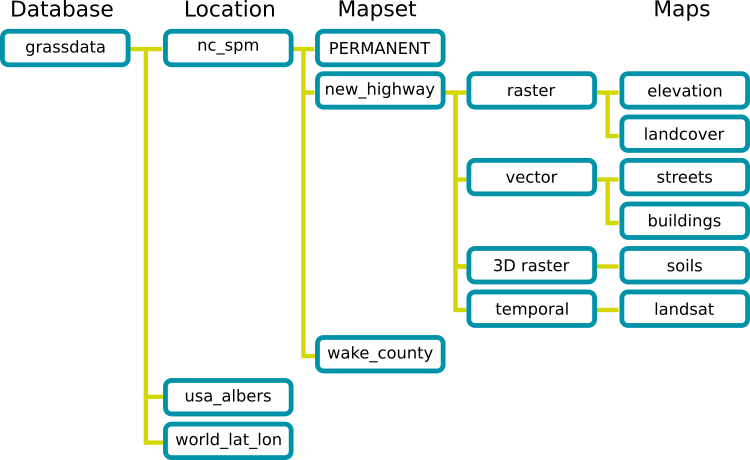
GRASS usa una terminología y estructura de bases de datos específica (GRASS GIS Spatial Database) que es importante entender para trabajar con GRASS GIS de manera efectiva. Necesita crear una nueva Localización e importar los datos requeridos a esta Localización. En lo que sigue vamos a revisar la terminología importante y dar direcciones paso a paso de cómo descargar y colocar sus datos en el lugar correcto.
- Una Base de datos espacial GRASS GIS (base de datos GRASS) consiste de un directorio con Localizaciones específicas (proyectos) donde los datos (capas/mapas) son almacenados.
- Una Localización es un directorio de datos relacionados a una ubicación geográfica y a un proyecto. Todos los datos dentro de una Localización tienen el mismo sistema de coordenadas de referencia.
- Un Directorio de mapas (Mapset) es una colección de mapas dentro de una Localización, contiene datos relacionados a una tarea específica, usuario o proyecto menor.
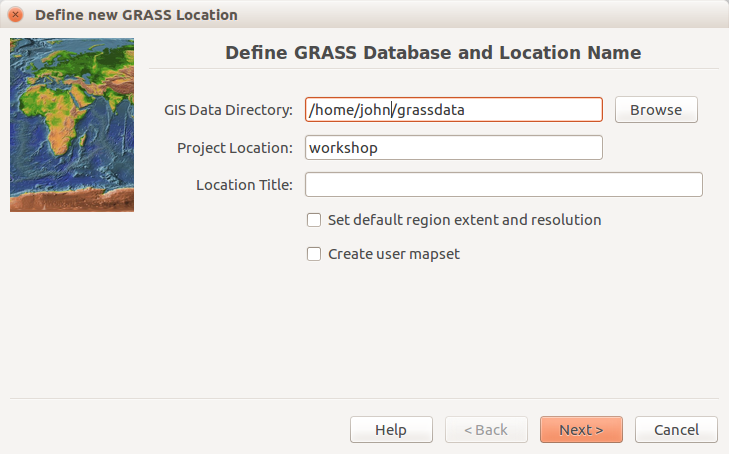
Inicie GRASS GIS, debe aparecer una pantalla de inicio. A menos que ya tenga un directorio llamado grassdata en su directorio de Documentos (en MS Windows) o en su directorio home (en Linux), cree uno. Puede usar el botón de Explorar y el cuadro de diálogo en la pantalla de inicio de GRASS GIS para hacer eso.
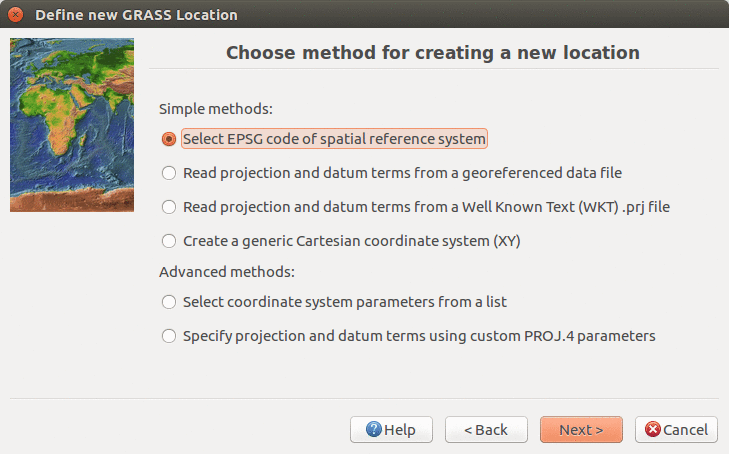
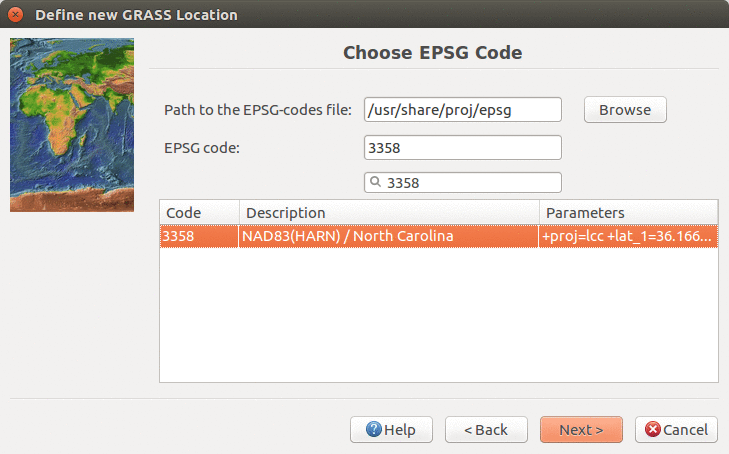
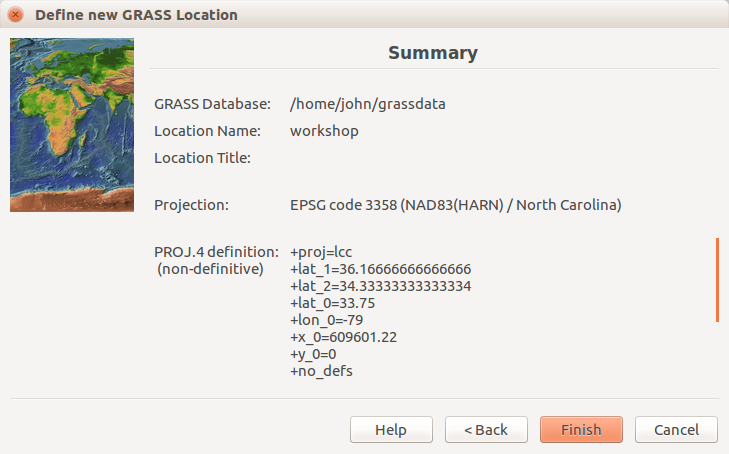
Va a crear una nueva Localización de su proyecto con el CRS (sistema de coordenadas de referencia) NC State Plane Meters cuyo código EPSG es 3358. Abra el Ayudante de localizaciones con el botón Nuevo en la parte izquierda de la pantalla de bienvenida. Seleccione el nombre de la nueva Localización, seleccione el método EPSG y el código 3358.
Cuando el Ayudante haya finalizado, la nueva localización aparecerá enlistada en la pantalla de inicio. Seleccione la nueva localización y el directorio de mapas PERMANENT y presione Iniciar sesión de GRASS.
-
Estructura de la base de datos espacial de GRASS GIS
-
Cuadro de diálogo de inicio de GRASS GIS 7.2
-
Iniciar el Ayudante de Localizaciones e ingrese el nombre de la nueva Localización
-
Seleccionar método para describir el CRS
-
Encontrar y seleccionar EPSG 3358
-
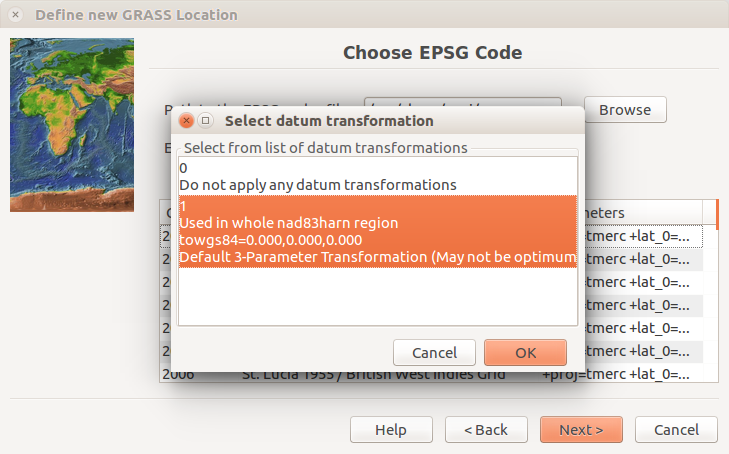
Confirmar el uso de la transformación de datum predeterminada
-
Revisar página de resumen y conrfirmar
Note que el directorio de trabajo actual es un concepto que es independiente de la base de datos GRASS, la Localización y el Directorio de mapas discutidos más arriba. El directorio de trabajo actual es el directorio en el cual cualquier programa (no solo GRASS GIS) escribe y lee archivos a menos que se le de una ruta para el archivo. El directorio de trabajo actual puede ser modificado desde la GUI usando Configuraciones → Entorno de trabajo de GRASS → Cambiar entorno de trabajo o desde la consola usando el comando cd. Esto es ventajoso cuando se está usando la línea de comandos y trabajando con el mismo archivo muchas veces, lo cuál suele suceder cuando se trabaja con datos lidar. Podemos cambiar el directorio al directorio en el cual se descargó el archivo LAS. En caso de que no cambiemos el directorio, podemos proveer la ruta completa al archivo. Note que la línea de comandos y la GUI tienen cada una su propia configuración del directorio de trabajo, así que se deben cambiar de manera independiente.
Importando los datos
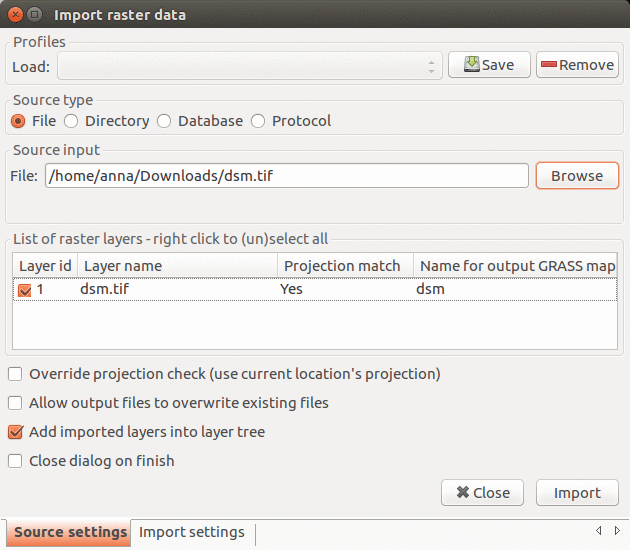
En este paso vamos a importra los datos a GRASS GIS. En el mení Archivo - Importar datos ráster seleccione Formatos comunes para importación y en el cuadro de diálogo explore para encontrar el archivo con la ortofoto, cambie el nombre a ortho.tif, y de click en el botón Importar. Todas las capas importadas deben ser añadidas a la GUI automáticamente, si no es así, añádalas manualmente. Las nubes de puntos será importadas más tarde de una manera diferente como parte del análisis.
-
Importar datos ráster: seleccione el archivo con la ortofoto y cambie el nombre a ortho
El comando equivalente es:
r.import input=nc_orthophoto_1m_spm.tif output=ortho
Región computacional
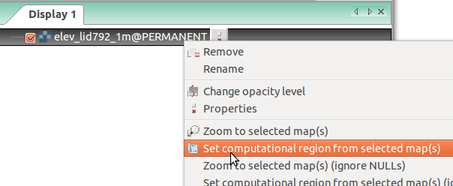
Antes de usar el módulo para calcular un nuevo mapa ráster, debemos definir adecuadamente la región computacional. Todos los cálculos con ráster deben ser realizados en una extensión especificada y con una resolución determinada.
La región computacional es un concepto importante en GRASS GIS. En GRASS una región computacional puede ser definida para realizar cálculos en áreas pequeñas o subregiones para probar el análisis o analizar regiones específicas, como por ej. unidades administrativas. Vamos a dar algunos puntos para mantener en mente cuando use la función de la región computacional.
- definida por la extensión y resolución ráster
- aplica a todas las operaciones ráster
- persiste entre sesiones de GRASS, puede ser diferente para distintos Directorios de mapas
- ventajas: mantiene consistencia en resultados, evita el recorte, para cálculos computacionalmente demantantes se puede definir una región de extensión menor y revisar si el resultado es correcto para luego realizar en toda la región o área de estudio.
- corra
g.region -po en el menú Configuraciones - Región - Mostrar región para ver la configuración actual de la región
-
Concepto de región computacional: un ráster con una extensión grade (azul) se muestra junto con otro más pequeño (verde). La región computacional (rojo) se define para que sea igual al ráster pequeño, así los cálculos estarán limitados a la región que ocupa el ráster menor aún cuando la entrada sea el ráster mayor. (No se muestra en la imagen: también la resolución, no solo la extensión, es igual a la resolución del ráster menor.)
-
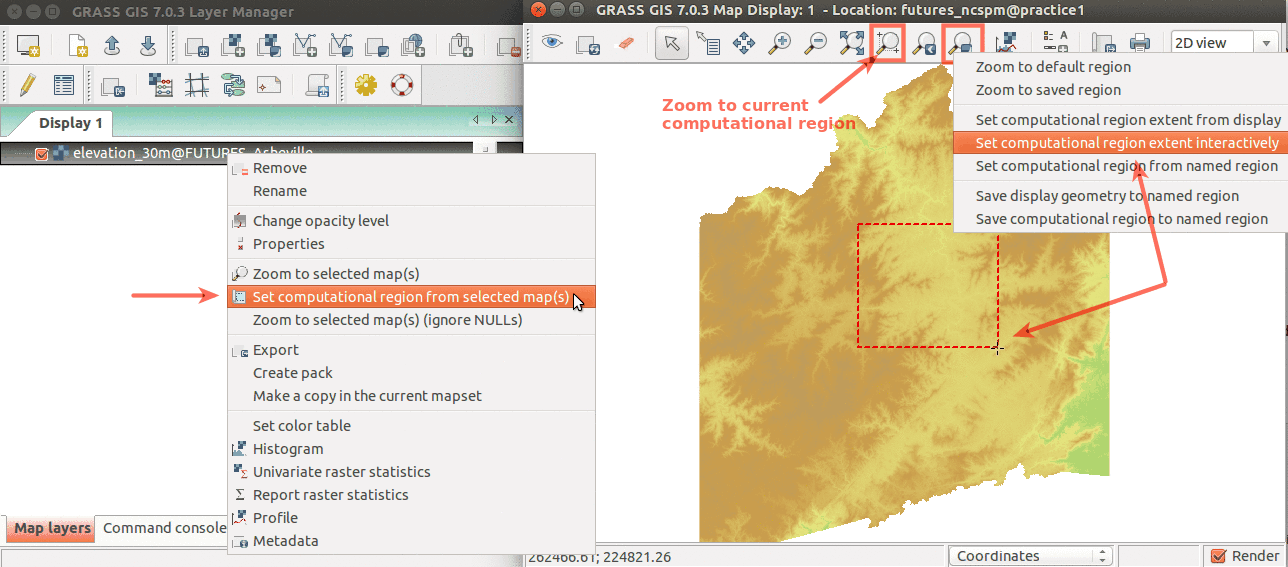
Modos sencillos para definir la región computacional desde la GUI. A la izquierda, definir la región para que sea igual a un mapa ráster. A la derecha, seleccionar la opción señalada y luego defina la región dibujando un rectángulo.
-
Definir la región computacional (extensión y resolución) para que sea igual a un ráster (pestaña de Capas en el Administrador de capas)
Los valores numéricos de la región computacional pueden ser revisados usando:
g.region -p
Luego de ejecutar el comando, obtendrá algo como esto:
norte: 220750 sur: 220000 oeste: 638300 este: 639000 nsres: 1 eores: 1 rows: 750 columnas: 700 celdas: 525000
La región computacional puede ser definida usando un mapa ráster:
g.region raster=ortho -p
La resolución se puede definir de manera separada usando el parámetro res del módulo g.region. Las unidades son las mismas de la Localización actual, en nuestro caso metros. Esto se puede hacer en la pestaña Resolución del díalogo de g.region o en la línea de comandos del siguiente modo (usando también la bandera -p para mostrar los nuevos valores:
g.region res=3 -p
La nueva resolución puede modificarse ligeramente en este caso para ajustar a la región, que no estamos cambiando. Sin embargo, a menudo queremos que la resolución sea igual a los valores que proveemos y estamos de acuerdo con una pequeña modificación a la extensión. Para eso está la bandera -a. De otro modo, si es importante alinear las celdas con un ráster específico, el parámetro align puede ser usado para alinear con el ráster (sin importar su extensión).
El siguiente ejemplo va a usar la extensión del ráster llamado ortho, usando una resolución de 5 metros, modifica la extensión para alinearlo a la resolución de 5 m y muestra los valores de la configuración de la región computacional:
g.region raster=ortho res=5 -a -p
Módulos
La funcionalidad de GRASS está disponible a través de módulos (herramientas, funciones). Existe en GRASS una convención de nombrar a los módulos de acuerdo a su función, de la siguiente manera:
| Prefijo | Función | Ejemplo |
|---|---|---|
| r.* | procesamiento ráster | r.mapcalc: álgebra de mapas |
| v.* | procesamiento vectorial | v.surf.rst: interpolación de superfícies |
| i.* | procesamiento de imágenes | i.segment: segmentación de imágenes |
| r3.* | procesamiento ráster 3D | r3.stats: estadísticas de ráster 3D |
| t.* | procesamiento de datos temporales | t.rast.aggregate: agregación temporal |
| g.* | administración general de datos | g.remove: remueve mapas |
| d.* | pantalla | d.rast: mostrar mapa ráster |
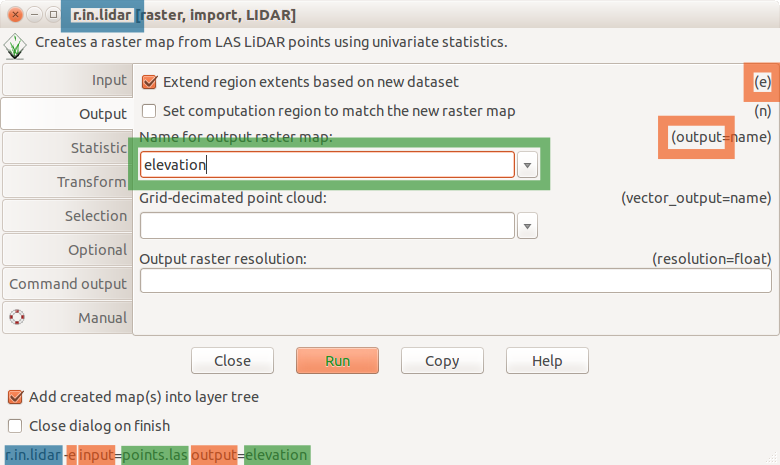
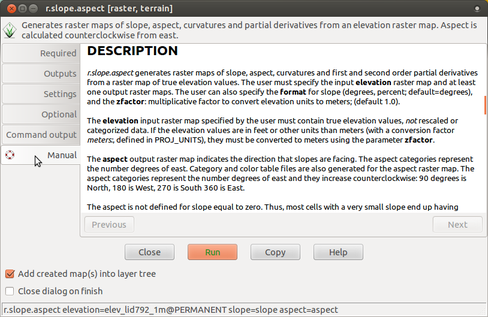
Estos son los grupos de módulos principales. Hay algunos otros que son específicos. Note también que algunos módulos tienen varios puntos dentro del nombre. Esto sugiere otro agrupamiento. Por ejemplo, los módulos que inician con v.net. tienen que ver con análisis de redes vectoriales. El nombre del módulo ayuda a entender su función, por ejemplo v.in.lidar inicia con v así que tiene que ver con mapas vectoriales, el nombre continúa con in lo que indica que el módulo es para importar datos a la Base de datos espacial de GRASS GIS y finalmente lidar indica que tiene que ver con nubes de puntos lidar.
-
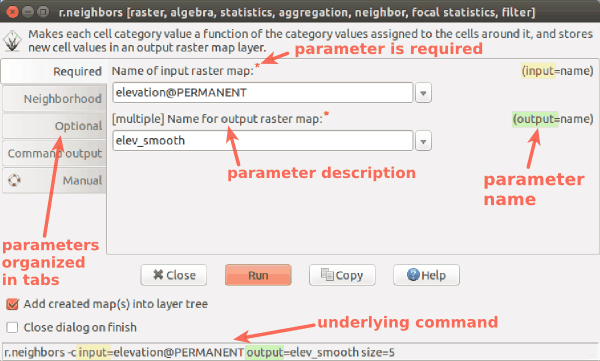
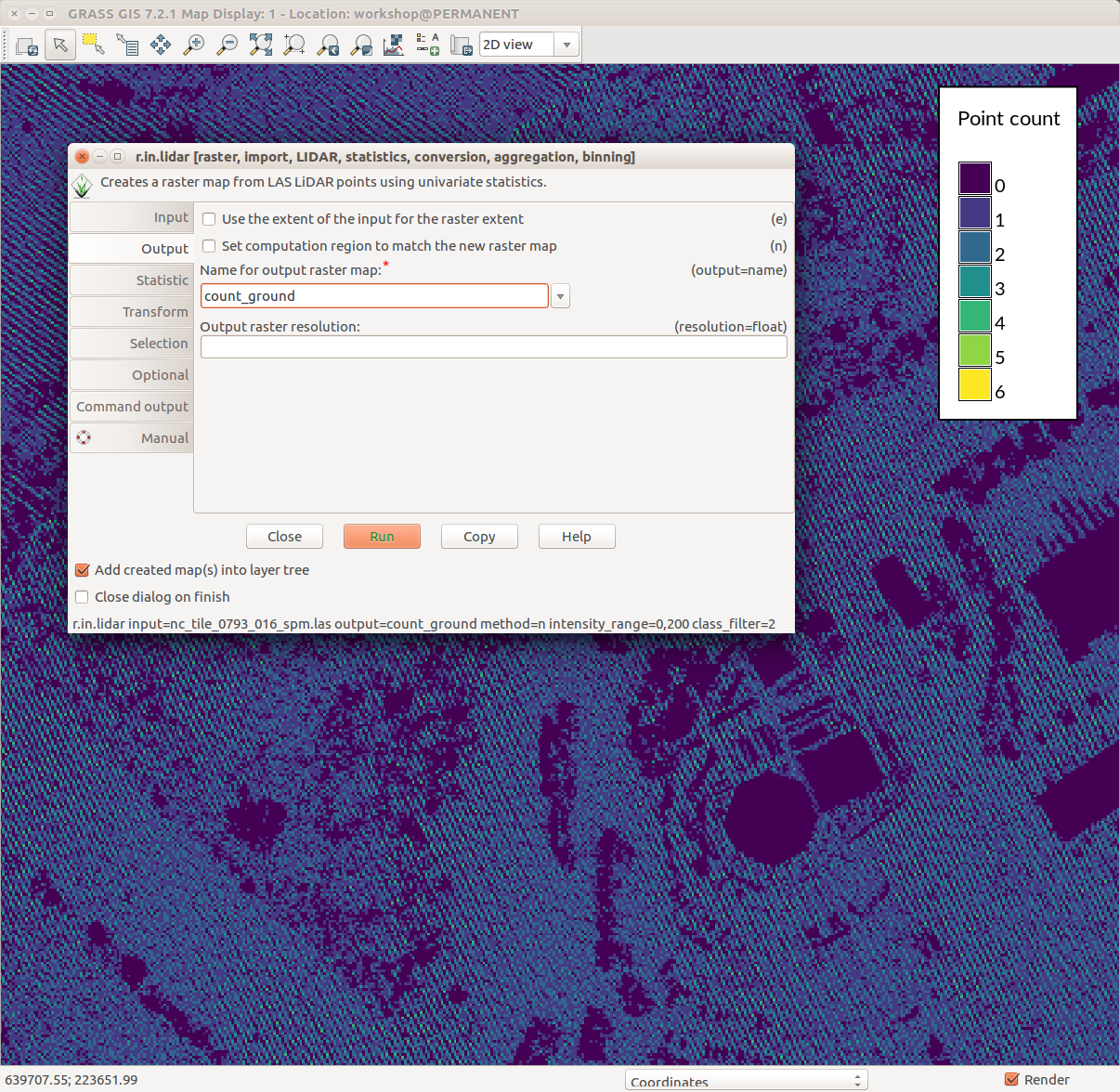
Diálogo del comando r.in.lidar con la pestaña de Salida activa y señalado el nombre del módulo (azul), las opciones y banderas (rojo) y la opción de valores (verde)
-
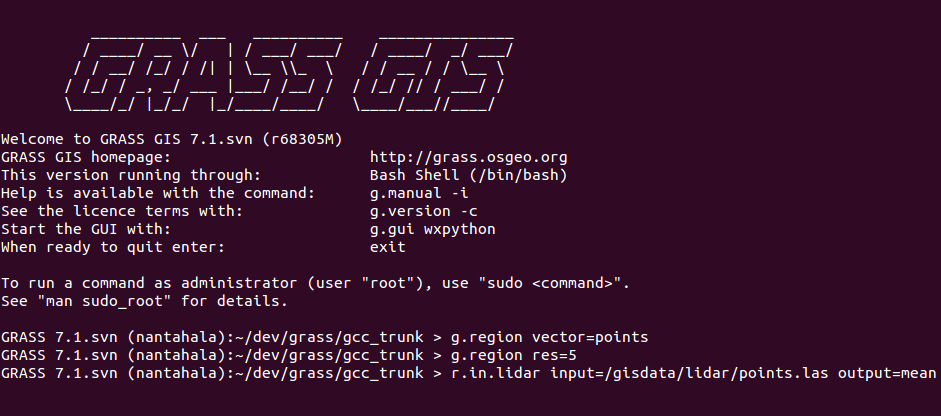
Ejemplo del comando r.in.lidar en la Bash señalado el nombre del módulo (azul), opciones y banderas (rojo) y opciones de valores (verde)
-
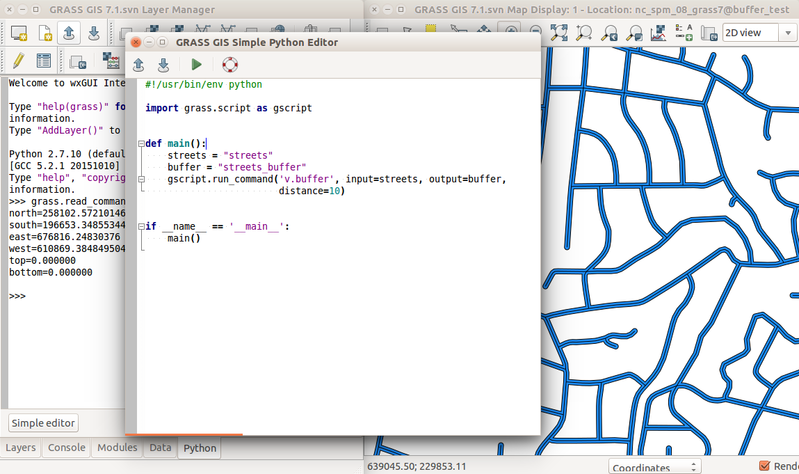
Ejemplo del comando r.in.lidar en Python señalado el nombre del módulo (azul), opciones y banderas (rojo) y opciones de valores (verde) y el comando import (gris)
-
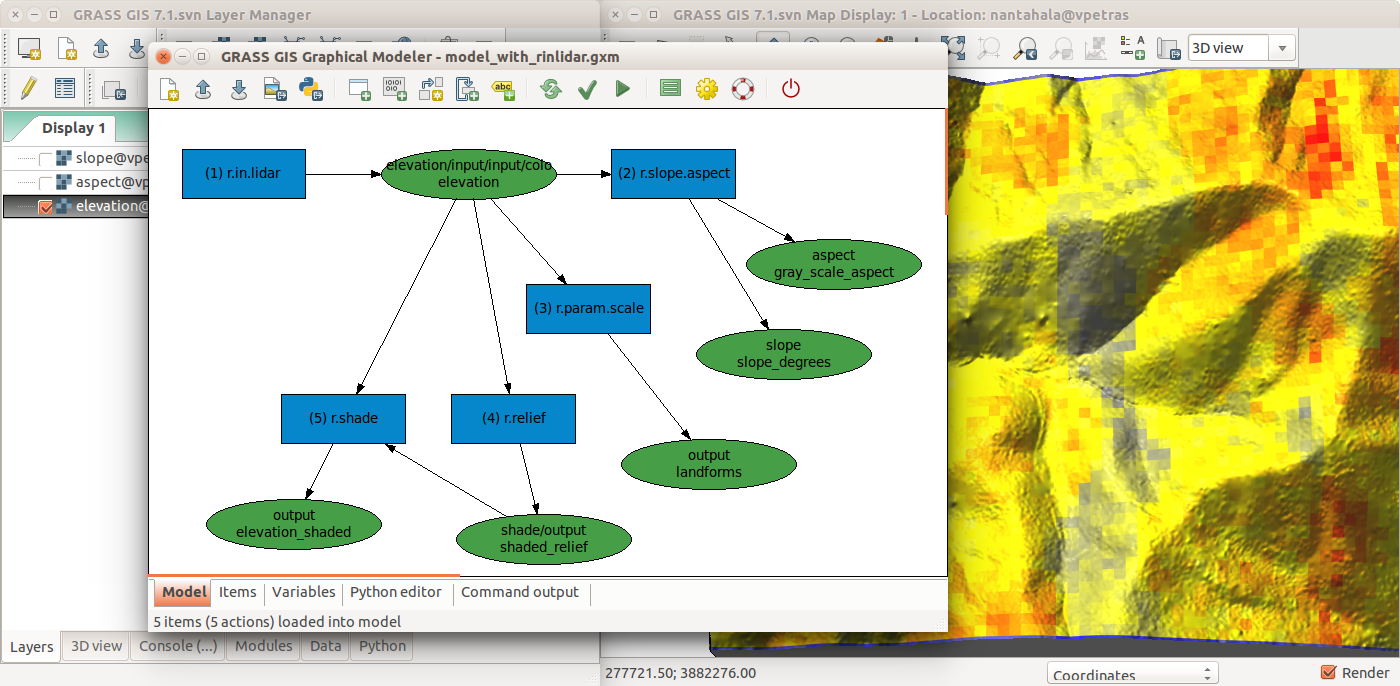
Modelador gráfico de GRASS GIS
-
Pestaña de Consola en el Administrador de capas para correr comandos y abrir diálogos de módulos
-
Interfaz de línea de comandos en un sistema de terminal
-
Muestra de un diálogo de un módulo
-
Buscar un módulo en la pestaña de Módulos
-
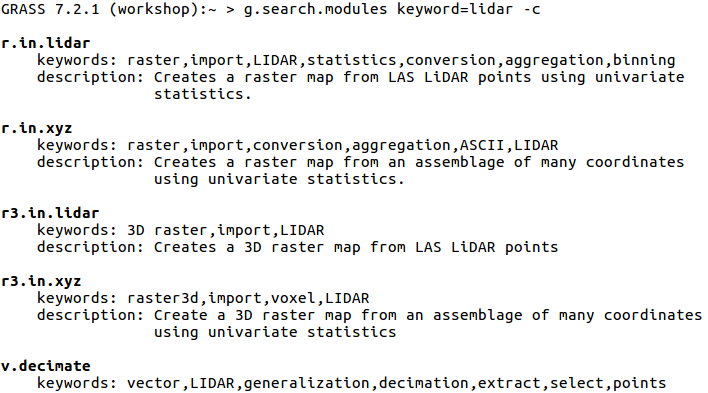
Buscar un módulo usando una búsqueda avanzada con g.search.modules
Una de las ventajas d eGRASS es la diversidad y cantidad de módulos que le permiten analizar los diferentes aspectos espaciales y temporales. GRASS GIS tiene más de 500 módulos distintos en la distribución núcleo y más de 300 módulos de extensión (addon) que pueden ser usados para preparar y analizar datos. La siguiente tabla enlista algunos de los principales módulos para análisis de nubes de puntos.
| Módulo | Función | Alternativas |
|---|---|---|
| r.in.lidar | binning into 2D raster, estadísticas | r.in.xyz, v.vect.stats, r.vect.stats |
| v.in.lidar | importar, decimar | v.in.ascii, v.in.ogr, v.import |
| r3.in.lidar | binning into 3D raster | r3.in.xyz, r.in.lidar |
| v.out.lidar | exporta nubes de puntos | v.out.ascii, r.out.xyz |
| v.surf.rst | interpola superficies a partir de puntos | v.surf.bspline, v.surf.idw |
| v.lidar.edgedetection | detección de bordes (ground and object) | v.lidar.mcc, v.outlier |
| v.decimate | decimar (adelgazar, thin) una nube de puntos | v.in.lidar, r.in.lidar |
| r.slope.aspect | parámetros topográficos | v.surf.rst, r.param.scale |
| r.relief | cálculo de relieve sombreado | r.skyview, r.local.relief |
| r.colors | administrador de tabla de colores de ráster | r.cpt2grass, r.colors.matplotlib |
| g.region | administrador de resolución y extensión | r.in.lidar, GUI |
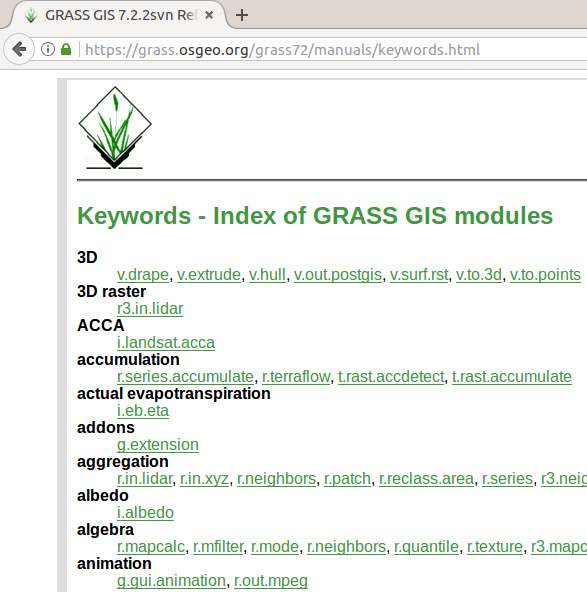
Los módulos y su descripción con ejemplos pueden ser encontrados en la documentación. La documentación es incluida en la instalación local y también disponible online.
-
Lista de palabras clave (claves, tags) en la documantación online
-
Página de manual de un módulo disponible también desde el diálogo del módulo
Introducción básica a la interfaz de Python
La manera más sencilla de ejecutar el código de Python que usa los paquetes de GRASS GIS es usar el Editor simple de Python integrado a GRASS GIS (accesible desde la barra de herramientas o la pestaña Python en el Administrador de capas). Otra opción es usar su editor de texto favorito y luego correr el script de GRASS GIS usando el menú principal Archivo → Lanzar script.
-
Editor simple de Python integrado en GRASS GIS
-
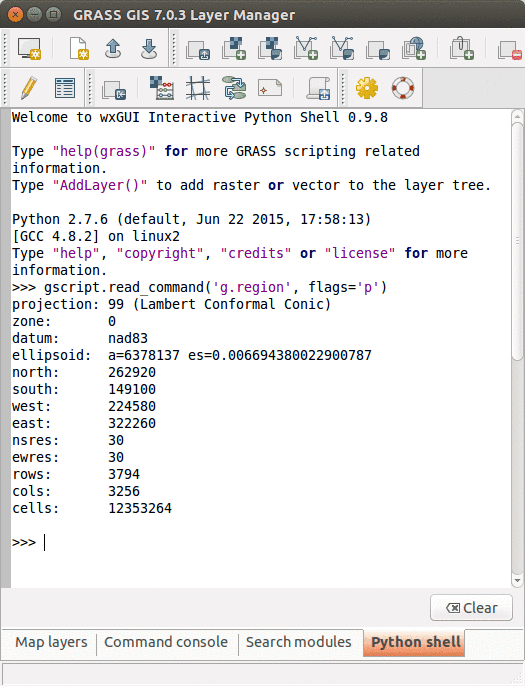
Pestaña Python con una interfaz interactiva de consola de Python
Vamos a usar el Editor simple de Python para correr los comandos. Puede abrirlo desde la pestaña Python. Cuando abra el Editor Simple de Python, verá un corto código. Empieza importando la librería GRASS GIS Python Scripting Library:
import grass.script as gscript
En la función principal llamamos g.region para ver la configuración actual de la región computacional:
gscript.run_command('g.region', flags='p')
Note que la sintáxis es similar a la de la línea de comandos (g.region -p), solo que la bandera se especifica como un parámetro. Ahora podemos correr el script presionando el botón Correr en la barra de herramientas. En el administrador de capas obtenemos la salida de g.region.
En este ejemplo, definimos la extensión y resolución a la capa ráster ortho, esto se hará usando g.region raster=ortho en la línea de comandos.
Para usar el run_command para definir la región computacional, reemplace el comando previo g.region con la siguiente línea:
gscript.run_command('g.region', raster='ortho')
La librería GRASS GIS Python Scripting Library provee funciones para llamar módulos de GRASS dentro de scripts de Python como subprocesos. Todas las funciones están en un paquete llamado grass y las funciones más comunes están en el paquete grass.script que es comúnmente importado import grass.script as gscript. Las funciones más frecuentemente usadas incluyen:
- script.core.run_command(): usada para módulos cuya salida es un vectorial o ráster y no se espera salida de texto
- script.core.read_command(): usada cuando interesa la salida de texto, que se obtiene como un texto de Python (string, cadena)
- script.core.parse_command(): usada con módulos que producen texto como pares clave=valor y que son procesados automáticamente como diccionarios de Python
- script.core.write_command(): para módulos que esperan ingreso de texto, ya sea de un archivo o de la entrada estándar
Ahora usamos parse_command para obtener las estadísticas como un diccionario Python
region = gscript.parse_command('g.region', flags='g')
print region['ewres'], region['nsres']
Los resultados mostrados son las resoluciones en la dirección E-O y N-S.
En este ejemplo, llamamos el módulo g.region. Típicamente, los scripts (y los módulos GRASS GIS) no cambian la región computacional y generalmente no necesitan ni leerla. La región computacional puede ser definida antes de correr el script así el script puede ser usado con diferentes parámetros de la región.
La librería también provee muchas funciones wrapper para los módulos que se usan con frecuencia como por ejemplo: script.raster.raster_info() (wrapper para r.info), script.core.list_grouped() (una de las wrappers para g.list), y script.core.region() (wrapper para g.region).
Cuando queremos correr el script de nuevo, necesitamos ya sea remover o crear datos de antemano usando g.remove o necesitamos decir a GRASS GIS que sobreescriba los datos existentes. Esto se puede hacer añadiendo overwrite=True como un argumento adicional a la función para cada módulo o podemos hacerlo de manera global usando os.environ['GRASS_OVERWRITE'] = '1' (requiere ejecutar previamente: import os).
Finalmente, puede haber notado que la primer línea del script dice #!/usr/bin/env python. Esto es lo que Linux, Mac OS y otros sistemas similares usan para determinar el intérprete a usar. Si obtiene algo como [Errno 8] Exec format error, esta línea probablemente esté incorrecta o faltante.
Decidir si usar la GUI, la línea de comandos, Python o el Jupyter Notebook
- GUI: se puede combinar con la línea de comandos (recomendado, especialmente para principiantes, combinar con copiar y pegar a la consola)
- línea de comandos: puede ser combinado con la GUI, la mayoría de las instrucciones siguientes serán para la línea de comando (pero puede ser fácilmente transferidas a la GUI o a Python), tanto la línea de comandos como la pestaña de Consola en la GUI funcionan bien.
- Python: necesita cambiar la sintáxis a Python; use fragmentos de este notebook estático notebook
- Jupyter Notebook localmente: es recomendado solamente si está usando OSGeoLive o si está en Linux o está familiarizado con Jupyter desde GRASS GIS; descargue el notebook desde github.com/wenzeslaus/Notebook-for-processing-point-clouds-in-GRASS-GIS
- Jupyter Notebook online: link distribuido por el instructor (opción de respaldo si otras fallan)
Binning de una nube de puntos
-
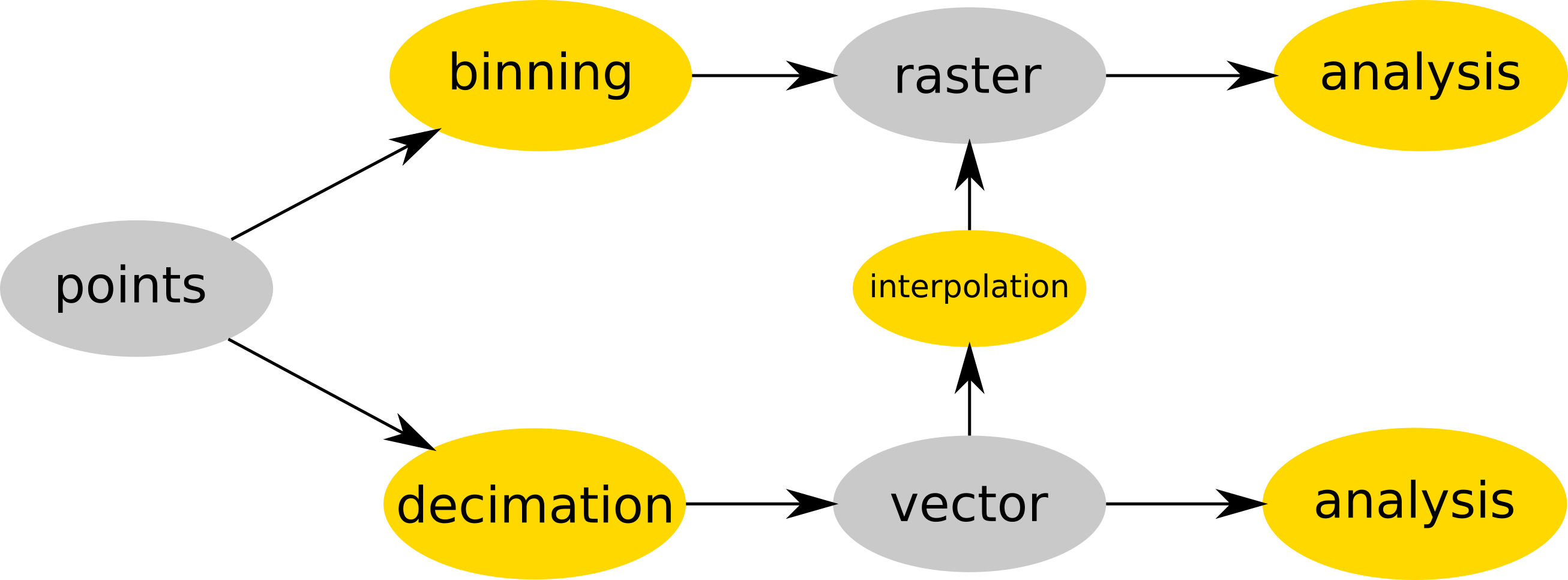
La nube de puntos es o almacenada (binned) en un ráster (ej. r.in.lidar) y luego analizada como ráster u opcionalmente decimada v.in.lidar), convertida a un vectorial (usando v.in.lidar) y luego interpolada (ej. v.surf.rst). Datos en gris, procesos en amarillo.
-
En el caso básico, el almacenado de los puntos en un ráster 2D consiste en contar el número de puntos que cae en cada celda. El valore resultante es la cantidad de puntos por celda.
-
En general el almacenado (binning) involucra también los valores asociados con los puntos y el cálculo de estadísticos de esos valores. Aquí la media de la coordenada Z de los puntos de cada celda es calculado y guardado en el ráster. La celda sin puntos son NULL (NoData, Sin datos), se muestra en color blanco.
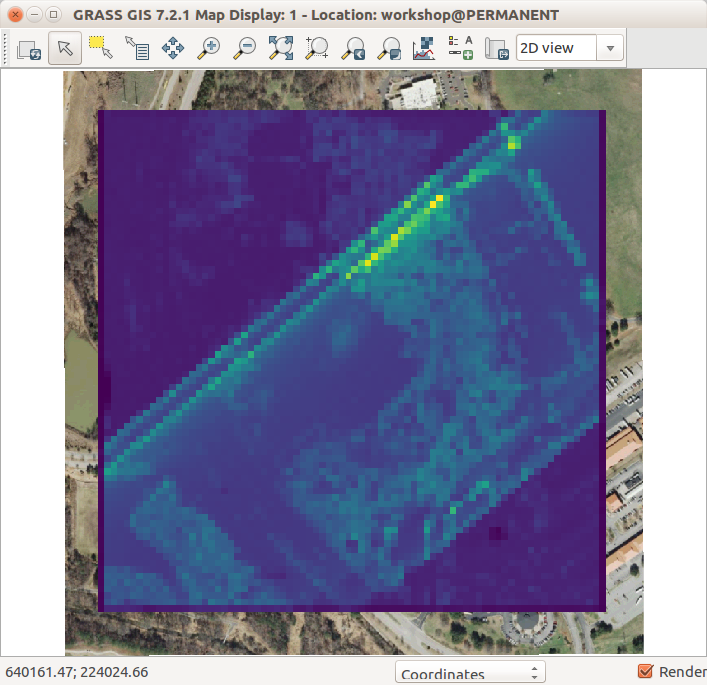
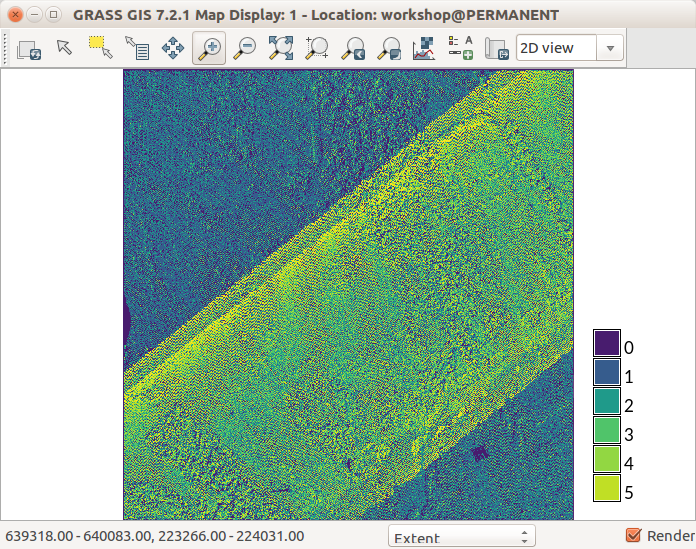
El modo más rápido de analizar las propiedades básicas de una nube de puntos es usar el almacenado (binning) y crear un mapa ráster. Vamos ahora a usar r.in.lidar para crear un ráster de conteo de putos (densidad de puntos). En este momento, no conocemos la extensión espacial de la nube de puntos, así que no podemos definir la región computacional, pero podemos decir al módulo r.in.lidar que determine la extensión primero y usarla para la salida usando la bandera -e. Estamos usando una resolución de grano grueso para maximizar la velocidad del proceso. Adicionalmente, la bandera -r define la región computacional para igualarla a la salida.
r.in.lidar input=nc_tile_0793_016_spm.las output=count_10 method=n -e -n resolution=10
Ahora podemos ver el patrón de distribución, pero examinemos también los números (en la GUI usando el botón derecho en el Administrador de capas y luego Metadatos o usando r.info directamente:
r.info map=count_10
Vamos a revisar rápidamente algunos de los valores usando la herramienta de consulta en el Visualizador de mapas. Dado que hay muchos puntos por celda, podemos hacer la resolución más fina:
r.in.lidar input=nc_tile_0793_016_spm.las output=count_1 method=n -e -n resolution=1
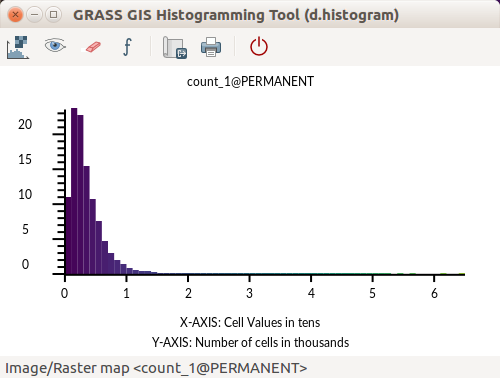
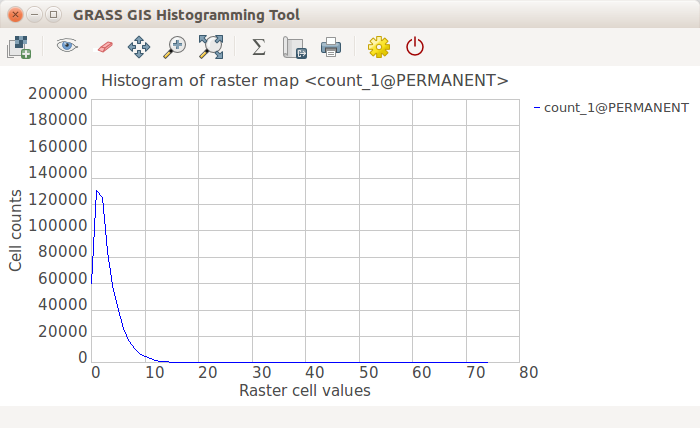
Vea la distribución de valores usando un histograma. Se accede al histograma desde el menú de contexto de la capa en el Administrador de capas, o desde la barra de herramientas del Visualizador de mapas con el botón Analizar mapa o usado el módulo d.histogram (d.histogram map=count_1).
-
Conteo de puntos por celda usando r.in.lidar con celdas de 10 m (
resolution=10) y bandera-e -
Herramienta de Histograma de GRASS GIS (basada en d.histogram)
-
Herramienta de Histograma GRASS GIS (basada en wxPython)
Ahora tenemos el número apropiado de puntos en la mayoría de las celdas y no hay artefactos de densidades alrededor de los bordes, así que podemos usar este ráster como base para la extensión y resolución que vamos a usar a partir de ahora.
g.region raster=count_1 -p
Sin embargo, la región tiene muchas celdas y algunas estarán vacías, especialmente cuando empecemos a filtrar la nube de puntos. Para tomar en cuenta esto, podemos modificar la resolución de la región computacional:
g.region res=3 -ap
Use el almacenado (binning) para obtener el modelo digital de superficie (DSM) (resolución y extesión tomados de la región computacional):
r.in.lidar input=nc_tile_0793_016_spm.las output=binned_dsm method=max
Para entender qué muestra el mapa, calcule los estadísticos usando r.report (Reportar estadísticas de ráster en el menú de contexto de la capa):
r.report map=binned_dsm units=c
Esto muestra que hay un dato discrepante (outlier) (alrededor de 600 m). Cambie el color de la tabla a un histograma ecualizado (bandera -e) para ver el contraste con el resto del mapa (usando la tabla de color viridis):
r.colors map=binned_dsm color=elevation -e
Vamos a revisar los datos discrepantes también usando el mínimo (que es method=min, ya usamos method=max para el DSM):
r.in.lidar input=nc_tile_0793_016_spm.las output=minimum method=min
Compare esto con el rango y la media de los valores en el ráster de suelo y decida cual es el rango permisible.
r.report map=minimum units=c
De nuevo, para ver los datos, podemos usar un histograma con una tabla de color ecualizada:
r.colors map=minimum color=elevation -e
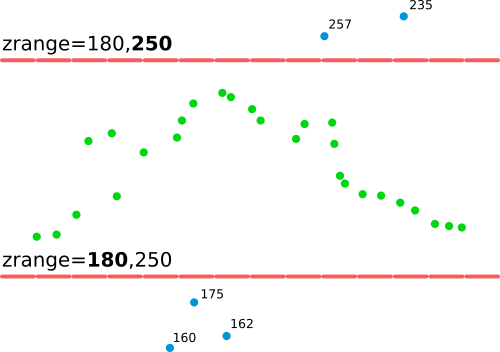
zrange filtra los puntos que no caen en el rango de valores Z especificadosAhora cuando sabemos cuales datos son discrepantes y cual es el rango de valores esperados (60-200 m parece ser un rango seguro pero amplio), use el parámetro zrange para filtrar cualquier posible punto discrepante (no confundir con el parámetro intensity_range) cuando se calcule un nuevo DSM:
r.in.lidar input=nc_tile_0793_016_spm.las output=binned_dsm_limited method=max zrange=60,200
Interpolación
Ahora vamos a interporlar un modelo digital de superficie (DSM) y para eso podemos incrementar la resolución para obtener tanto detalle como sea posible (podemos usar 0.5m, i.e. res=0.5, para mayor detalle o 2 m, i.e. res=2m -a, para mayor velocidad):
g.region raster=count_1 -p
Antes de interpolar, vamos a confirmar que la distribución espacial de los puntos permita interpolar de manera segura. Necesitamos usar el mismo filtro que vamos a usar para el DSM:
r.in.lidar input=nc_tile_0793_016_spm.las output=count_dsm method=n return_filter=first zrange=60,200
Primero revisamos los números:
r.report map=count_dsm units=h,c,p
Luego revisamos la distribución espacial. Para eso necesitamos usar el histograma con tabla de color ecualizada (la leyenda puede ser limitada solo a un rango específico de valores d.legend raster=count_interpolation range=0,5).
r.colors map=count_dsm color=viridis -e
Primero importamos el archivo LAS como puntos vectoriales, vamos a mantener solamente los primeros puntos de retorno y limitar la importación verticalmete para evitar usar los puntos discrepantes encontrados en los pasos previos. Antes de correrlo, deseleccionamos Añadir mapa(s) creados al árbol de capas en el diálogo de v.in.lidar si está usando la GUI.
v.in.lidar -bt input=nc_tile_0793_016_spm.las output=first_returns return_filter=first zrange=60,200
-
Opción Añadir mapa(s) creados al árbol de capas deseleccionada, en el diálogo de in v.in.lidar
-
Conteo de puntos en una resolución más fina con histograma con tabla de color viridis ecualizada y leyenda con un rango limitado
Luego interpolamos:
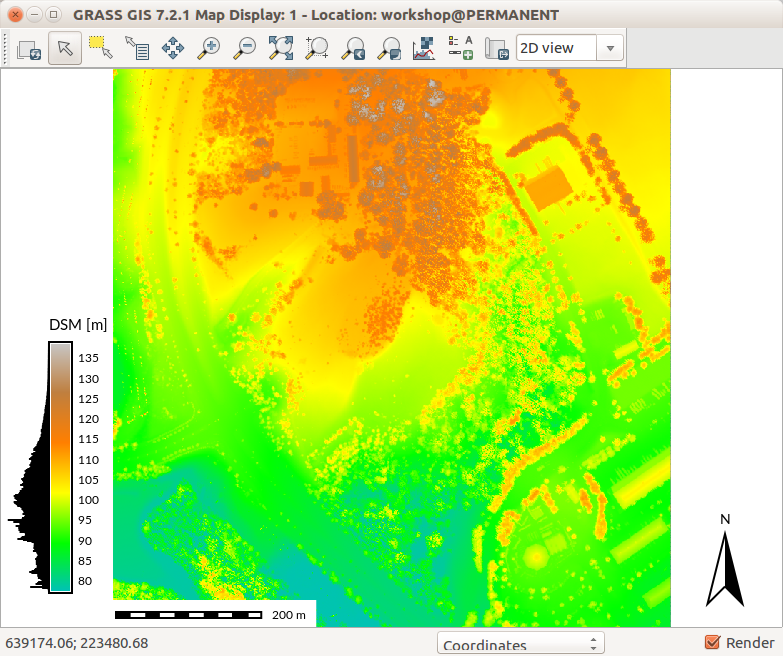
v.surf.rst input=first_returns elevation=dsm tension=25 smooth=1 npmin=80
Ahora podemos visualizar el DSM resultante calculando el relieve sombreado con r.relief o usando la vista 3D (ver secciones siguientes).
-
DSM con leyenda e histograma
Análisis de terreno
Definir la región computacional basada en un mapa ráster existente:
g.region raster=count_1 -p
Revisar si la densidad de puntos es suficiente (el suelo es la clase 2, resolución y extensión son tomados de la región computacional:
r.in.lidar input=nc_tile_0793_016_spm.las output=count_ground method=n class_filter=2 zrange=60,200
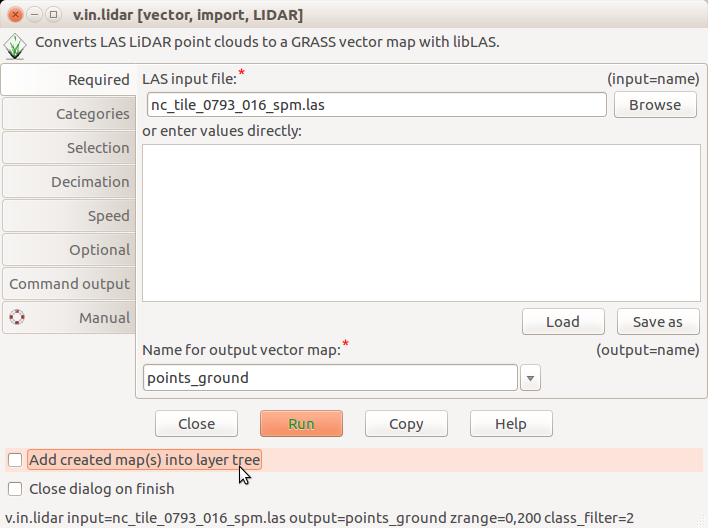
Importar puntos (la bandera -t deshabilita la creación de la tabla de atributos y la bandera -b deshabilita la construcción de topología; deseleccionar Anadir mapa(s) creados al árbol de mapas):
v.in.lidar -bt input=nc_tile_0793_016_spm.las output=points_ground class_filter=2 zrange=60,200
-
Visualizador de mapas con leyenda creada usando d.legend, diálogo de r.in.lidar, patrón de densidad de puntos en el fondo
-
Deseleccionar Añadir mapa(s) creados al árbol de capas en v.in.lidar
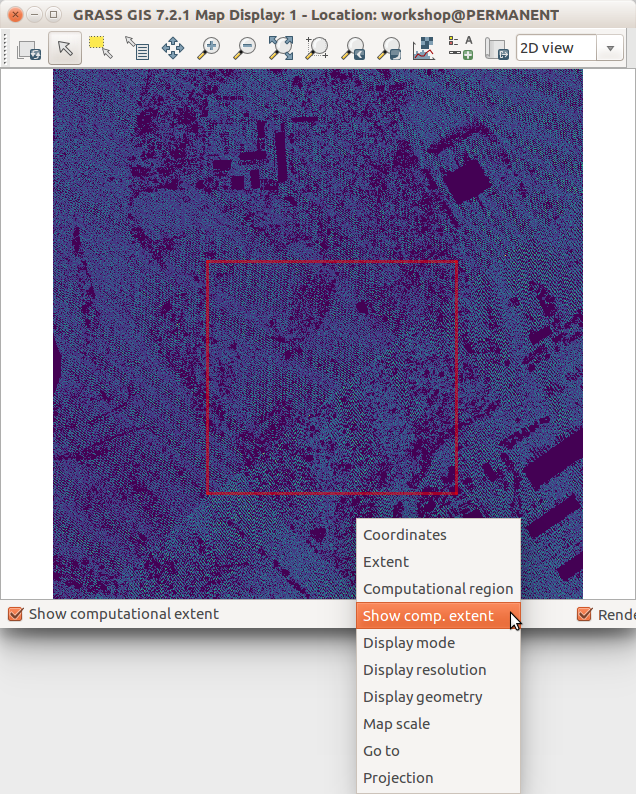
La interpolación tardará algún tiempo, así que vamos a definir la región computacional a un área menor para ahorrar tiempo antes de determinar cuales son los mejores parámetros para la interpolación. Podemos hacer esto en la GUI desde la barra de herramientas del Visualizador de mapas o usando las coordenadas:
g.region n=223751 s=223418 w=639542 e=639899 -p
-
Modos sencillos para definir la región computacional desde la GUI. A la izquierda, definir la región para que sea igual a un mapa ráster. A la derecha, seleccionar la opción señalada y luego defina la región dibujando un rectángulo.
-
Mostrar la extensión de la región computacional actual en el Visualizador de mapas
Ahora interpolamos la superficie del suelo usando spline regularizada con tensión (regularized spline with tension) (implementada en v.surf.rst) y al mismo tiempo derivamos la pendiente, aspecto y curvatura (el siguiente código es una linea larga):
v.surf.rst input=points_ground tension=25 smooth=1 npmin=100 elevation=terrain slope=slope aspect=aspect pcurvature=profile_curvature tcurvature=tangential_curvature mcurvatur=mean_curvature
Cuando examinamos los resultados, especialmente las curvaturas muestran un patrón que puede ser causado por algunos problemas con la colecta de la nube de puntos. Vamos a disminuir la tensión que va a causar la superficie para aumentar el suavizado y alejarse de los valores de los puntos. Dado que el mapa ráster ya existe, vamos a usar la bandera para sobreescribir, i.e. --overwrite en la línea de comandos, o en la GUI seleccionar para reemplazar los ráster existentes. Usamos la bandera --overwrite o solamente --o.
v.surf.rst input=points_ground tension=20 smooth=5 npmin=100 elevation=terrain slope=slope aspect=aspect pcurvature=profile_curvature tcurvature=tangential_curvature mcurvatur=mean_curvature --o
Cuando estemos satisfechos con el resultado, vamos de vuelta a la extensión deseada:
g.region raster=count_1 -p
Y finalmente interpolamos la superficie del suelo en toda la extensión:
v.surf.rst input=points_ground tension=20 smooth=5 npmin=100 elevation=terrain slope=slope aspect=aspect pcurvature=profile_curvature tcurvature=tangential_curvature mcurvatur=mean_curvature --o
Calculamos el relieve sombreado:
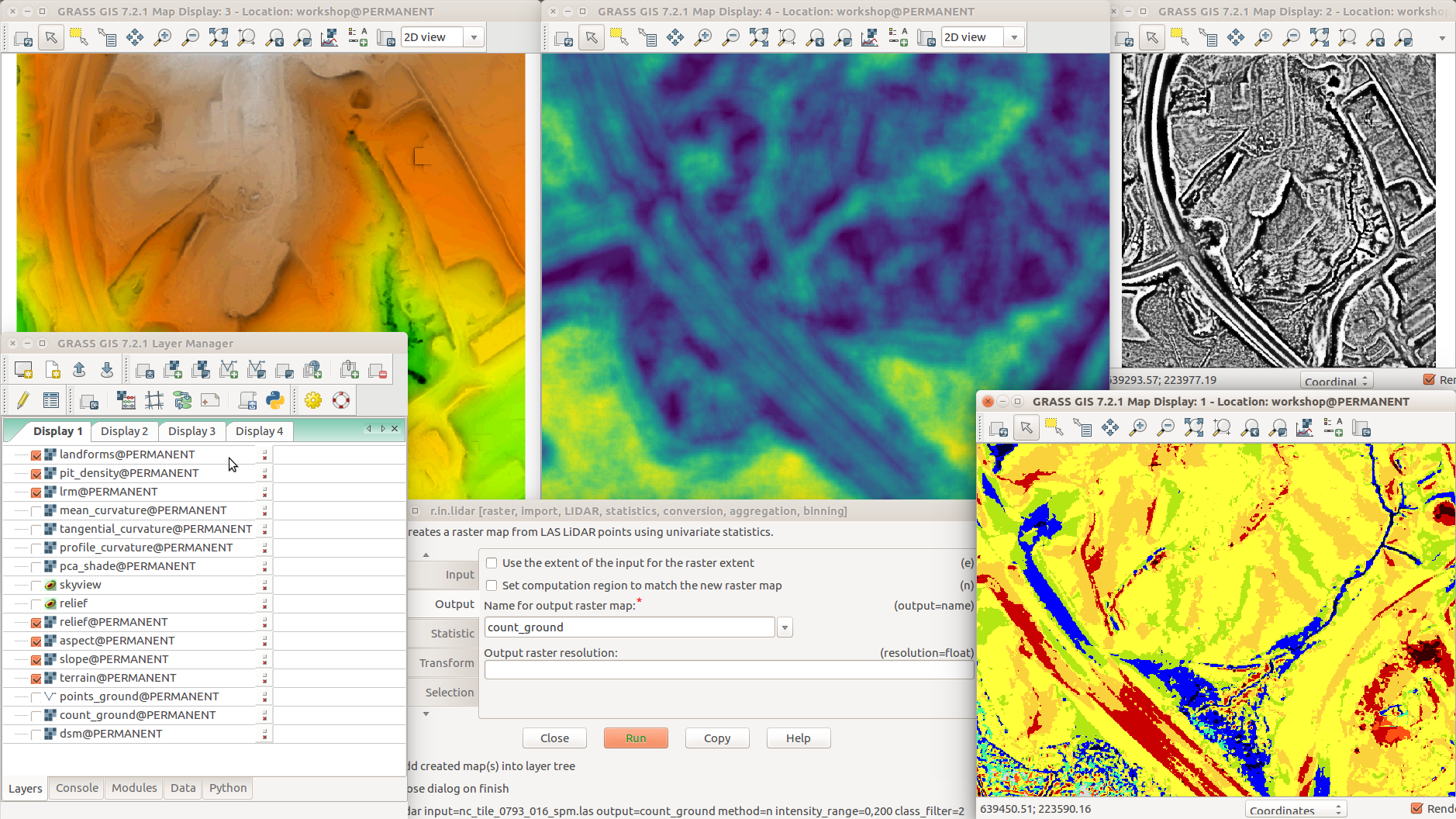
r.relief input=terrain output=relief
Ahora combinamos el ráster de relieve sombreado con el ráster de elevación. Esto se puede hacer de diferentes maneras. En la GUI cambiando la opacidad. Un mejor resultado se puede obtener con el módulo r.shade que combina dos rásters y crea uno nuevo. Finalmente, esto se puede hacer al vuelo son crear un nuevo ráster usando el módulo d.shade. El módulo puede ser usado desde la GUI a través de la barra de herramientas o en la pestaña Consola:
d.shade shade=relief color=terrain
Ahora, en lugar de usar r.relief, vamos a usar r.skyview:
r.skyview input=terrain output=skyview ndir=8 colorized_output=terrain_skyview
Combinar el terreno y vista aérea (skyview) al vuelo:
d.shade shade=skyview color=terrain
Visualización analítica basada en relieve sombreado:
r.shaded.pca input=terrain output=pca_shade
Modelo de relieve local:
r.local.relief input=terrain output=lrm shaded_output=shaded_lrm
Densidad de pozos:
r.terrain.texture elevation=terrain thres=0 pitdensity=pit_density
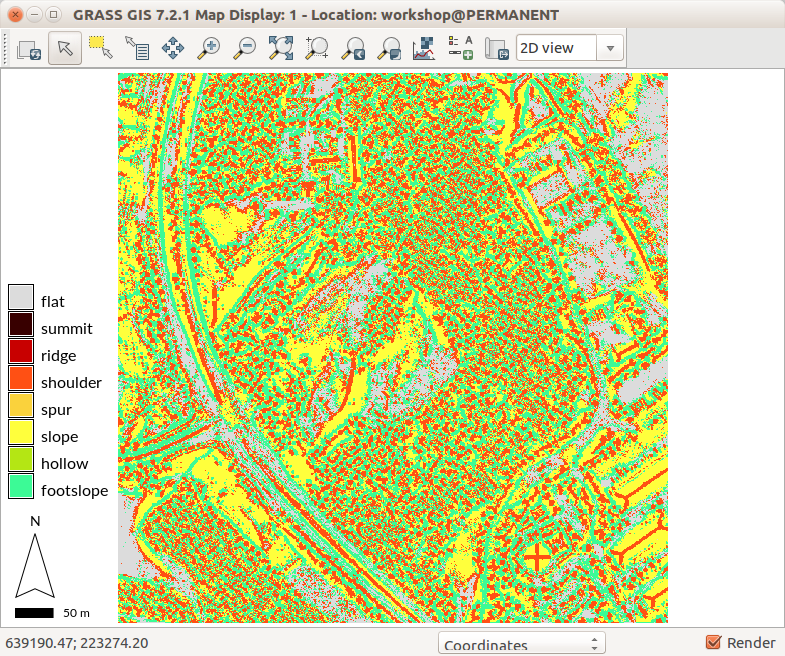
Finalmente, usamos la detección automática de formas de relieve (usando una ventana de búsqueda de 50 m):
r.geomorphon -m elevation=terrain forms=landforms search=50
-
Diferentes análisis de terreno y visualizaciones en múltiples Visualizadores de mapas
Análisis de vegetación

zrange filtra los puntos que no caen en el rango especificado de valores ZAunque para muchas aplicaciones relacionadas con la vegetación, una resolución más grues es más apropiada por que se necesitan más puntos para las estadísticas, vamos a usar solamente:
g.region raster=count_1
Vamos a usar todos los puntos (sin usar la clasificación de los puntos), pero con el filtrado de Z para obtener el rango de las alturas en cada celda:
r.in.lidar input=nc_tile_0793_016_spm.las output=range method=range zrange=60,200
Vamos a usar todos los puntos, pero con el filtro Z para obtener el rango de alturas en cada celda:
r.mapcalc "vegetation_by_range = if(range > 2, 1, null())"
O una menor resolución para evitar huecos (y posiblemente suavizado para rellenarlos).
-
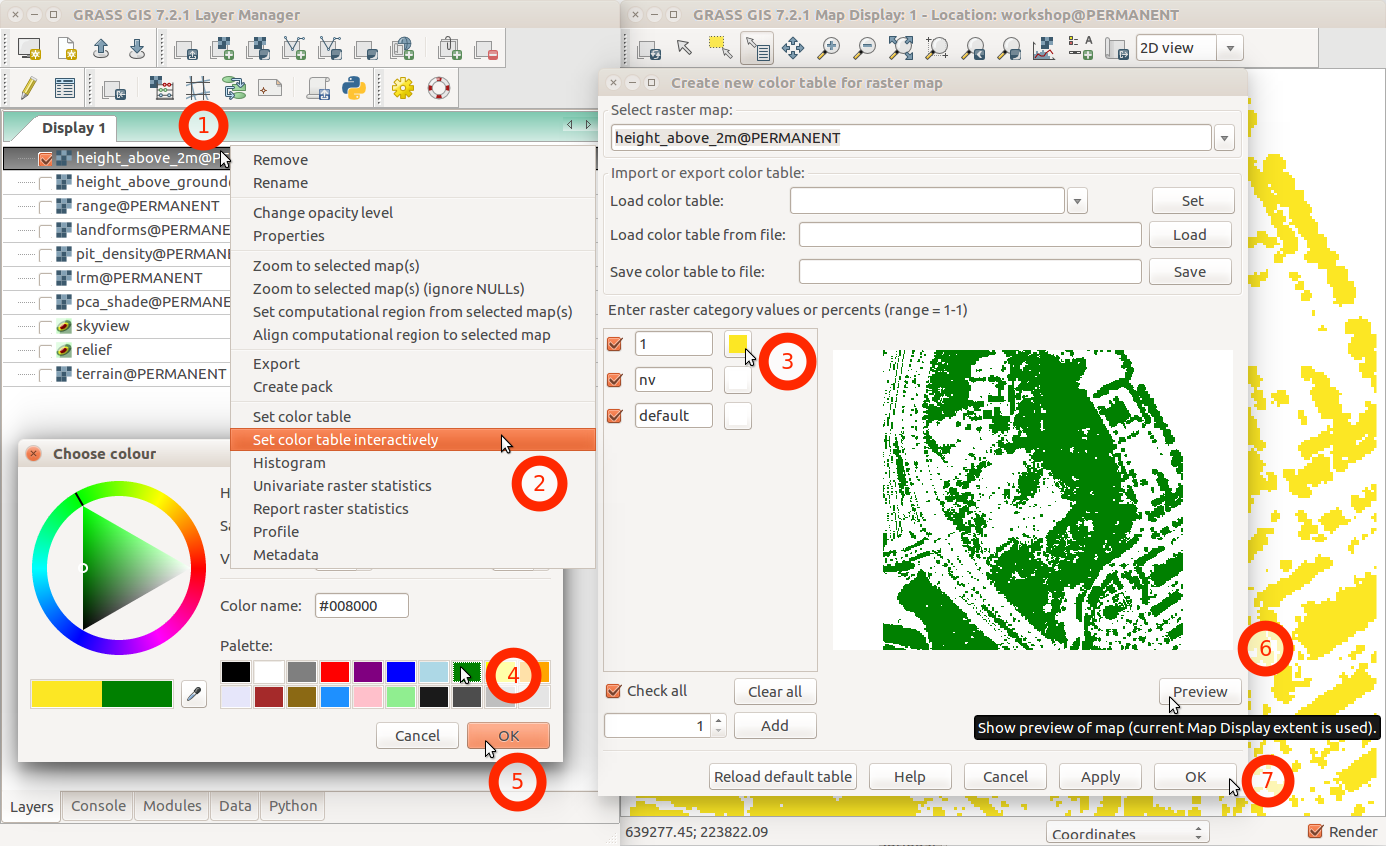
Cambiar la tabla de color para el mapa ráster desde el menú de contexto usando Definir tabla de colores de manera interactiva.
r.in.lidar -d input=nc_tile_0793_016_spm.las output=height_above_ground method=max base_raster=terrain zrange=0,100
En caso de que usemos if(height_above_ground > 2, height_above_ground, null()).
r.mapcalc "above_2m = if(height_above_ground > 2, 1, null())"
Usar r.grow para extender los parches y rellenar los huecos:
r.grow input=above_2m output=vegetation_grow
Consideramos el resultado para representar las áreas con vegetación. Vamos a agrupar (conectar) las celdas individuales en parches usando r.clump):
r.clump input=vegetation_grow output=vegetation_clump
Algunos de los parches son muy pequeños. Usando r.area removemos todos los parches menores que un determinado límite:
r.area input=vegetation_clump output=vegetation_by_height lesser=100
Ahora convertimos esas áreas a vectores:
r.to.vect -s input=vegetation_by_height output=vegetation_by_height type=area
Hasta ahora estamos usando la elevación de los puntos, ahora vamos a usar la intensidad.
La intensidad es usada por r.in.lidar cuando se le da la bandera -j (o -i):
r.in.lidar input=nc_tile_0793_016_spm.las output=intensity zrange=60,200 -j
Con esta mayor resolución, hay algunas celdas sin puntos, por lo que usamos r.fill.gaps para rellenar estas celdas (r.fill.gaps también suaviza el ráster como parte del proceso de rellenado de huecos)
r.fill.gaps input=intensity output=intensity_filled uncertainty=uncertainty distance=3 mode=wmean power=2.0 cells=8
La tabla de colores de Grey es más apropiada para la intensidad:
r.colors map=intensity_filled color=grey
Hay algunas áreas con intensidades muy altas, para visualmente explorar otras áreas, usamos la tabla de colores ecualizada con el histograma:
r.colors -e map=intensity_filled color=grey
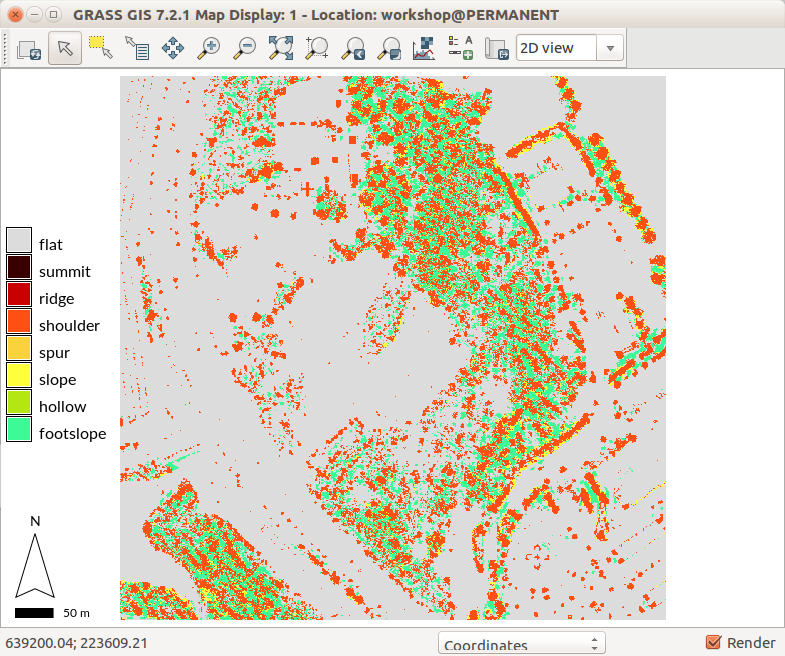
Vamos a usar r.geomorphon de nuevo, pero ahora con el DSM y diferente configuración. La siguiente configuración muestra las formas de los techos:
r.geomorphon elevation=dsm forms=dsm_forms search=7 skip=4
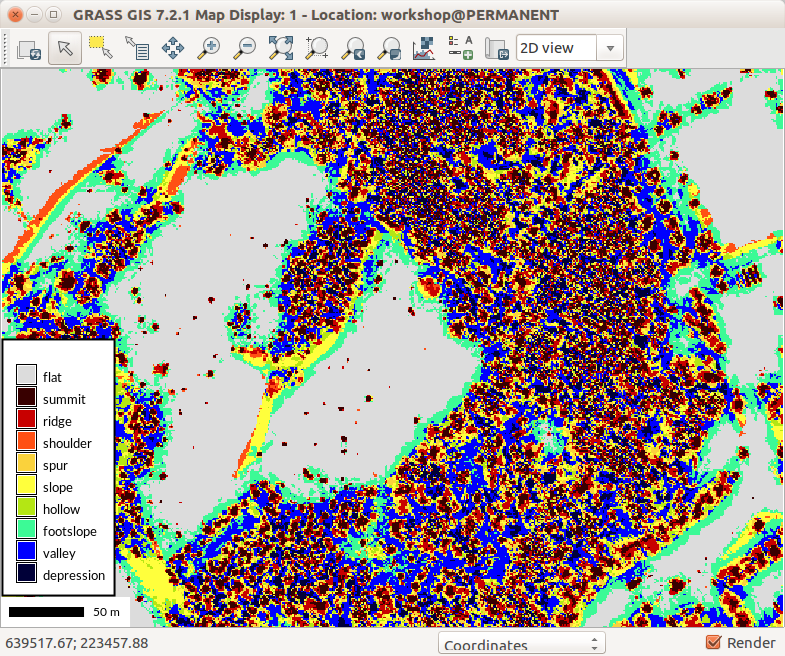
Diferentes configuraciones, espeecialmente un mayor límite de aplanado (flatness threshold) (parámetro flat) muestra áreas forestadas como combinaciones de (¿?) footslopes, slopes, and shoulders. Los árboles individuales se representan como shoulders.
r.geomorphon elevation=dsm forms=dsm_forms search=12 skip=8 flat=10 --o
Disminuyendo el parámetro skip disminuye la generalización y trae las cúspides que generalmente representan las copas de los árboles:
r.geomorphon elevation=dsm forms=dsm_forms search=12 skip=2 flat=10 --o
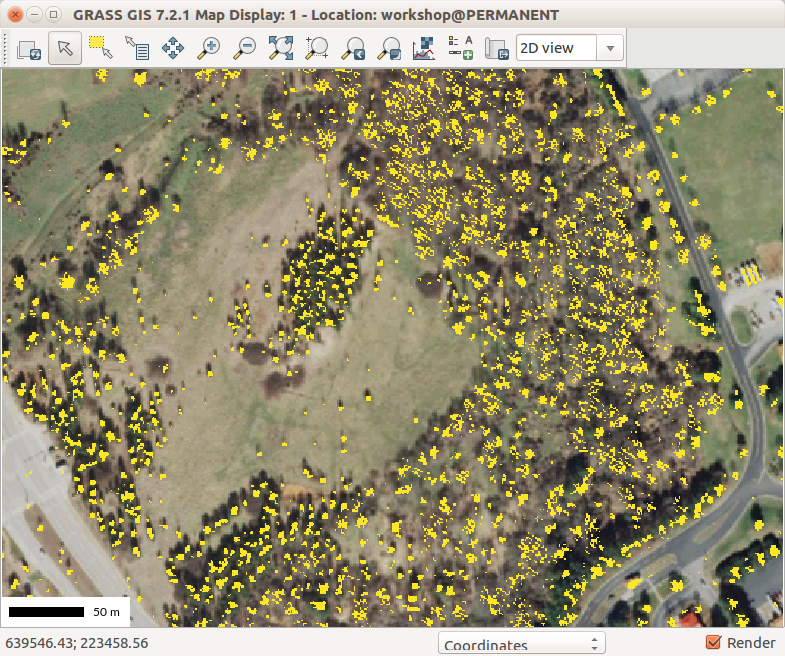
Ahora extraemos las cúspides usando r.mapcalc para álgebra de mapas con la expresión if(dsm_forms==2, 1, null()).
r.mapcalc "trees = if(dsm_forms==2, 1, null())"
-
Configuración de r.geomorphon que muestra bien las formas de los techos
-
Configuración diferente de r.geomorphon que muestra las áreas con vegetación
-
La configuración más común de r.geomorphon que resulta en una detallada descripción de las formas del DSM
-
Picos seleccionados desde r.geomorphon mostrando la posición de los árboles (copas de los árboles)
Tareas extra
Visualización 3D de rásters
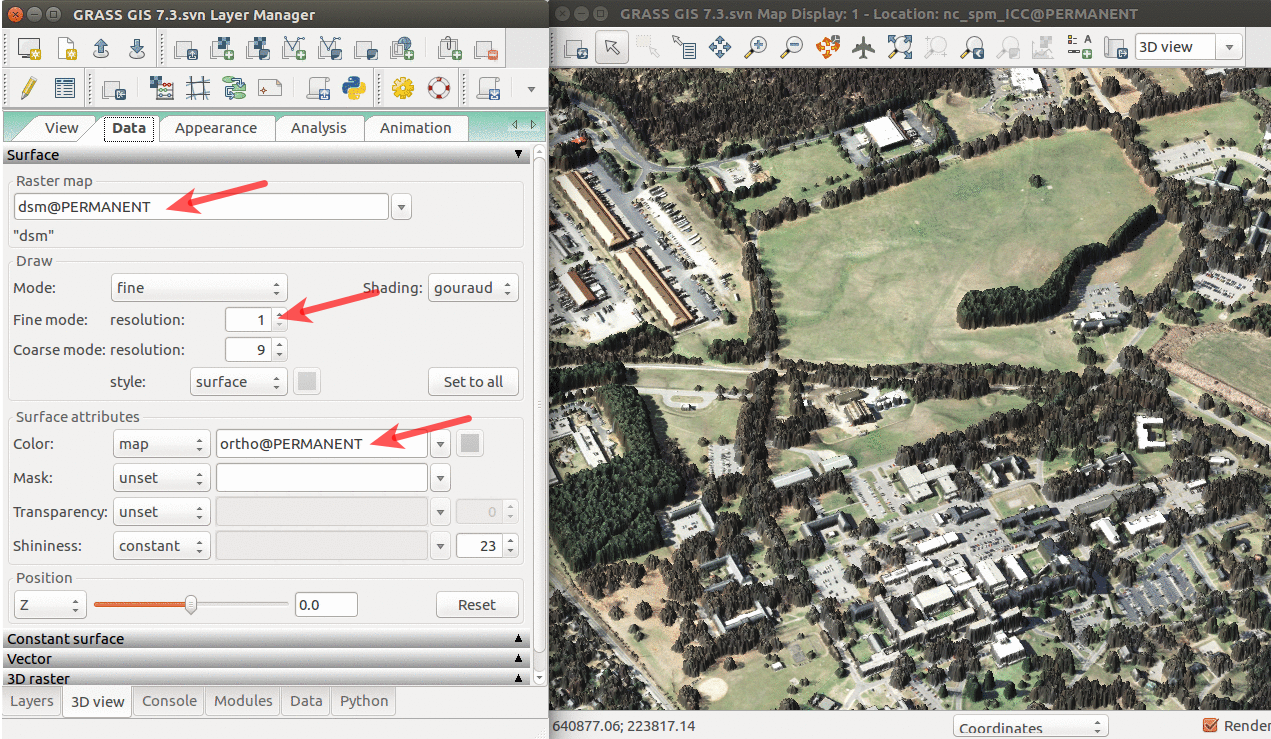
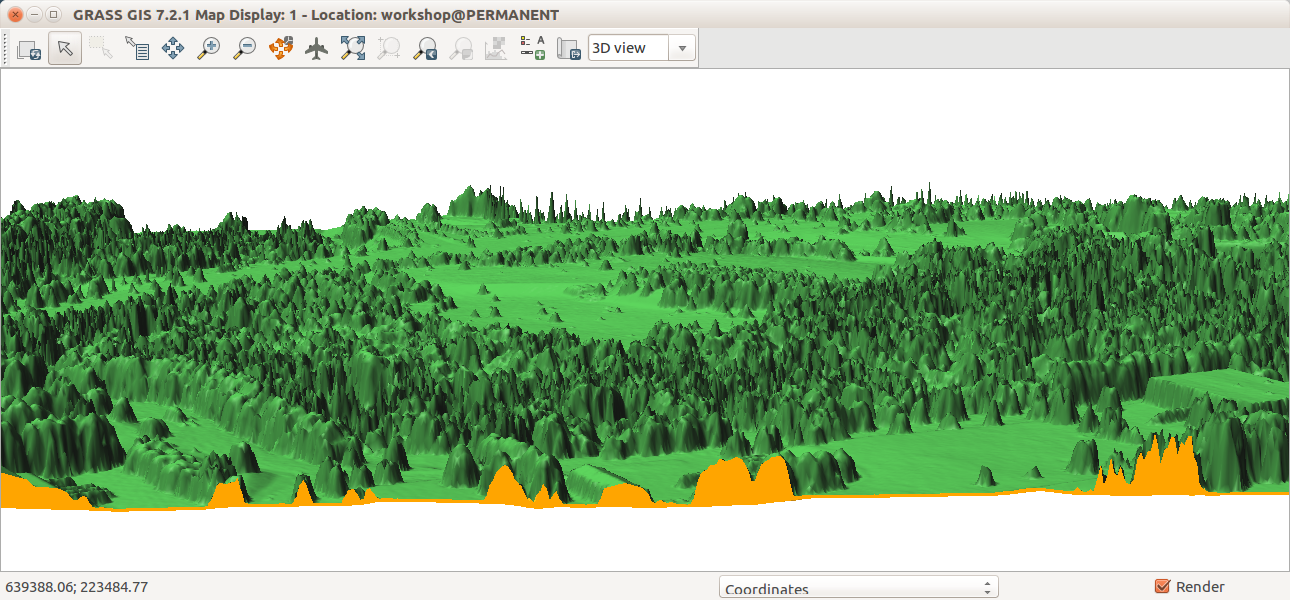
Podemos explorar nuestra área de estudio en la vista 3D (use la imagen de la derecha si necesita clarificación para los siguientes pasos):
- Añadir ráster dsm y deseleccionar o remover cualquier otra capa. Note que el deseleccionar (o remover) cualquier otra capa es importante porque cualquier otra capa cargada en Capas es interpretada como un suerficie en la vista 3D.
- Cambie a la vista 3D (en la esquina derecha del Visualizador de Mapas).
- Ajuste la vista (perspectiva, altura).
- En la pestaña Data, defina Modo de resolución fina a 1 y defina ortho como superficie de color (la ortofoto será mostrada sobre el DSM).
- Regrese a la pestaña Vista y explore las diferentes direcciones de vista usando el controlador verde.
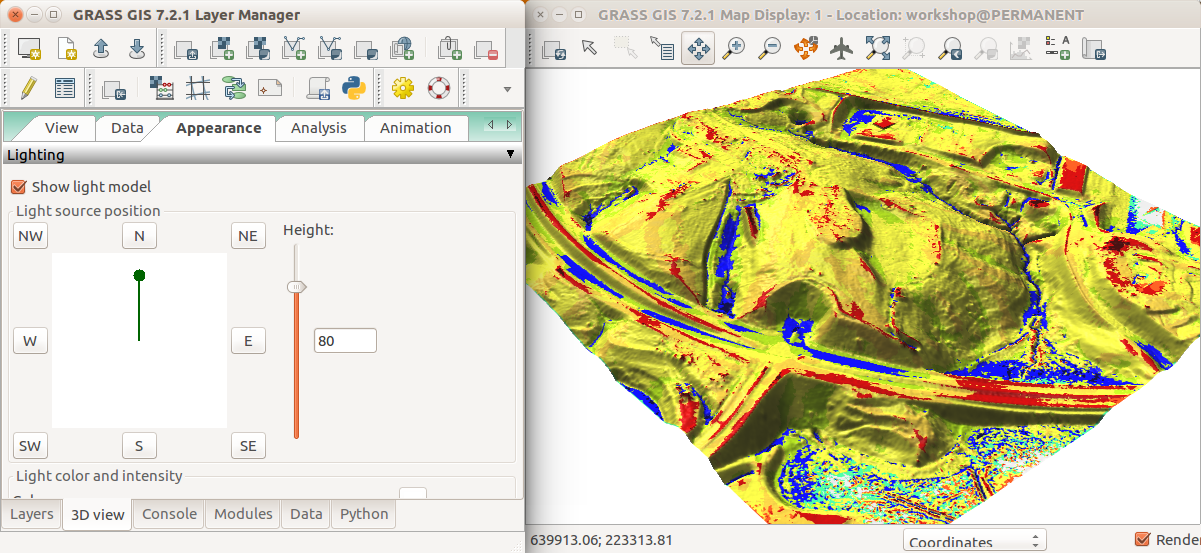
- Vaya a la pestaña Aparienciay cambie las condiciones de luz (mejor altura de la luz, cambio de dirección).
- Trate también usando el ráster landforms como la superficie de colores.
- Cuando termine, cambie de nuevo a la vista 2D.
-
Visualización 3D del DSM con la ortofoto superpuesta: las flechas muestran el nombre de la superficie, la resolución usada para visualizar, y el raster usado como color.
-
Formas de terreno sobre el DSM en 3D: la posición de la luz se cambia en la pestaña Apariencia
Visualización analítica de rásters en 3D
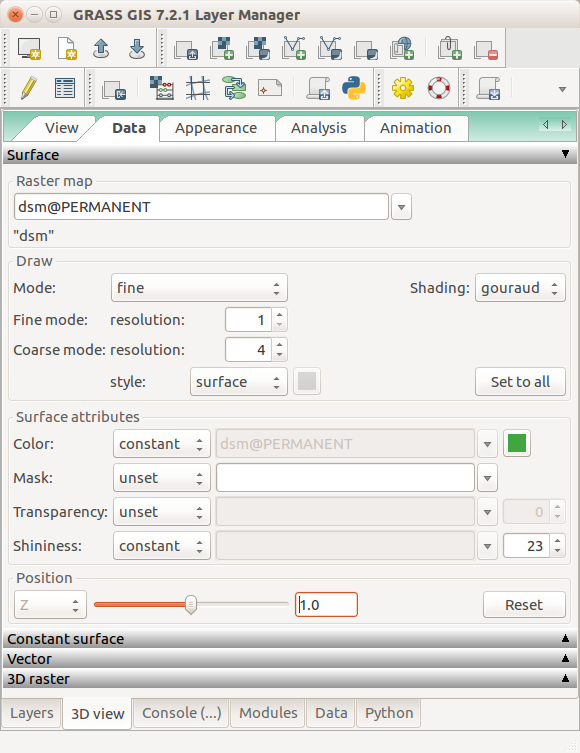
Podemos explorar nuestra área en vista 3D (use la imagen de la derecha si necesita clarificar los pasos siguientes):
- Añada los rásters terrain y dsm y deseleccione o remueva cualquier otra capa. (Cualquier capa en Capas será interpretada como superficie en la vista 3D.)
- Cambie a la vista 3D (en la esquina derecha del Visualizador de mapas).
- Ajuste la vista (perspectiva, altura). Defina la exageración-z a 1.
- En la pestaña Data, defina Modo de resolución fina a 1 para ambos rásters.
- Defina un color diferente para cada superficie.
- Para el dsm, defina la posición en la dirección Z a 1. Esta es la posición relativa a la posición actual del ráster. Esta pequeña compensación ayudará a ver la relación entre el terreno y la superficies del DSM.
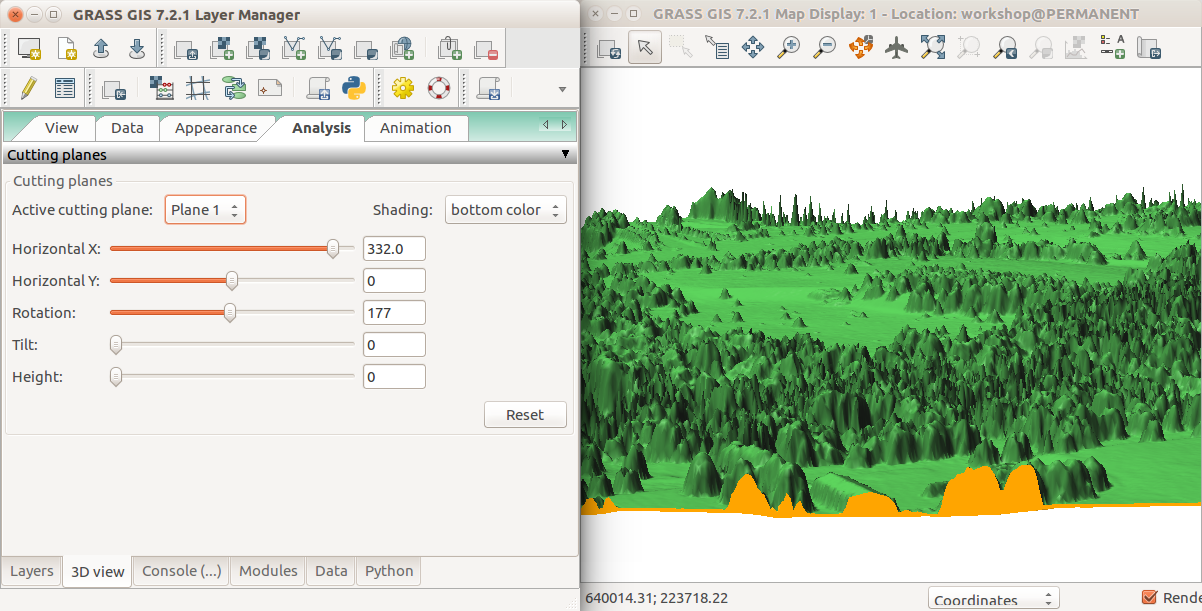
- Vaya a la pestaña Análisis y active el primer plano de corte. Defina Sombreado a bottom color para obtener el color de abajo para colorear el espacio entre el terreno y el DSM.
- Empiece a mover el plano para X, Y and Rotación.
- Vaya de nuevo a la pestaña de Vista para cambiar la vista.
- Cuando termine, cambie de nuevo a la vista 2D.
-
En la pestaña de Datos de la vista 3D, defina la resolución de ambas superficies. Escoja diferentes colores. Defina la posición (relativa) del DSM a 1, (por encima de la posición actual).
-
En la pestaña de Análisis de la vista 3D, active los planos de corte, defina el color de abajo (bottom color) e inicie a mover el plano para X, Y y rotación.
-
Para ver la parte más larga del transecto, amplíe la ventana horizontalmente.
Clasificar puntos de suelo y no suelo
Las nubes de puntos de UAV usualmente requieren clasificación de puntos de suelo (tierra solita) cuando queremos crear superficies de suelo.
Importar todos los puntos usando el comando v.in.lidar en la sección previa, a menos que ya los tenga. Dado que los metadatos acerca de la proyección son incorrectos use la bandera -o para saltar la revisión de consistencia de la proyección.
r.in.lidar -e -n -o input=nc_uav_points_spm.las output=uav_density_05 method=n resolution=0.5
Defina una región computacional menor para hacer todos los cálculos más rápidos (salte esto para hacer las operaciones en toda el área):
g.region n=219415 s=219370 w=636981 e=637039
Importe los puntos usando el módulo v.in.lidar pero limitando la extensión a la región computacional (-r flag), pero no construya la topología (bandera -b), no cree tampoco la tabla de atributos (bandera -t y no asigne categorías (ids) a los puntos, bandera -c). Hay más puntos que los que necesitamos para interpolar la resolución de 0.5 m, así que vamos a decimar (adelgazar, thin) la nube durante la importación, importando 75% de los puntos, usando preserve=4 (que usa una decimación basada en el conteo, que asume una distribución espacial uniforme de los puntos).
v.in.lidar -t -c -b -r -o input=nc_uav_points_spm.las output=uav_points preserve=4
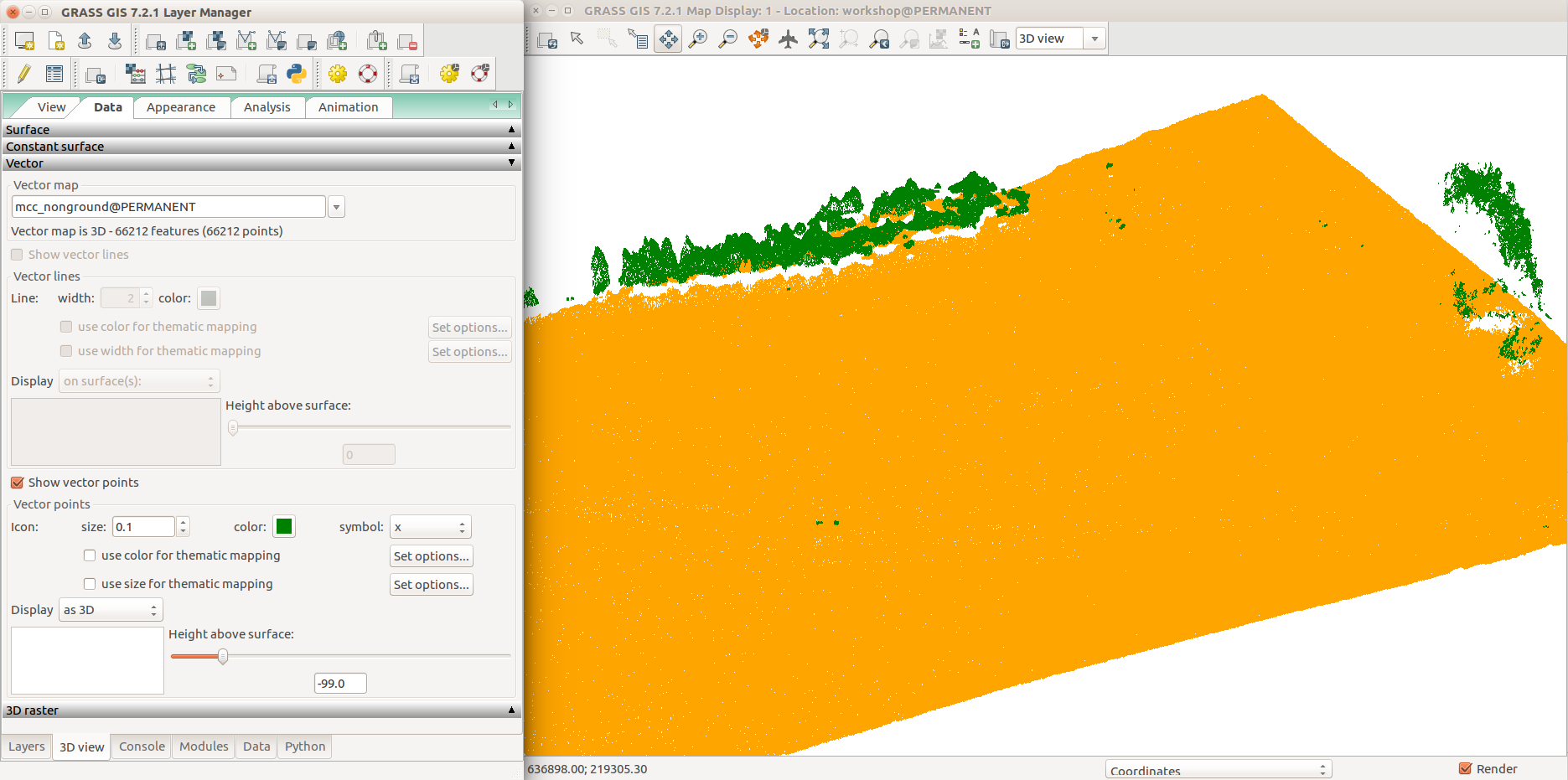
Luego use v.lidar.mcc para clasificar los puntos del suelo y los de no-suelo:
v.lidar.mcc input=points_all ground=mcc_ground nonground=mcc_nonground
Interpolar la superficie del suelo:
v.surf.rst input=mcc_ground tension=20 smooth=5 npmin=100 elevation=mcc_ground
Si está usando datos UAV, puede extraer los valores RGB de los puntos. Primero, importe los puntos de nuevo, pero esta vez con todos los atributos:
v.in.lidar -r -o input=nc_uav_points_spm.las output=uav_points preserve=4
Luego extraiga los valores RGB (guardados en columnas) en canales separados (ráters):
v.to.rast input=uav_points output=red use=attr attr_col=red v.to.rast input=uav_points output=green use=attr attr_col=green v.to.rast input=uav_points output=blue use=attr attr_col=blue
Y defina la tabla de colores a gris:
r.colors red color=grey r.colors green color=grey r.colors blue color=grey
Estos canales pueden ser usados para tareas de percepción remota. Ahora vamos a visualizarlos usando d.rgb:
d.rgb red=red green=green blue=blue
Si está usando datos lidar para esta parte, puede comparar el nuevo DEM con el conjunto de datos previo:n
r.mapcalc "mcc_lidar_differece = ground - mcc_ground"
Defina la tabla de colores:
r.colors map=mcc_lidar_differece color=difference
Visualización 3D denubes de puntos
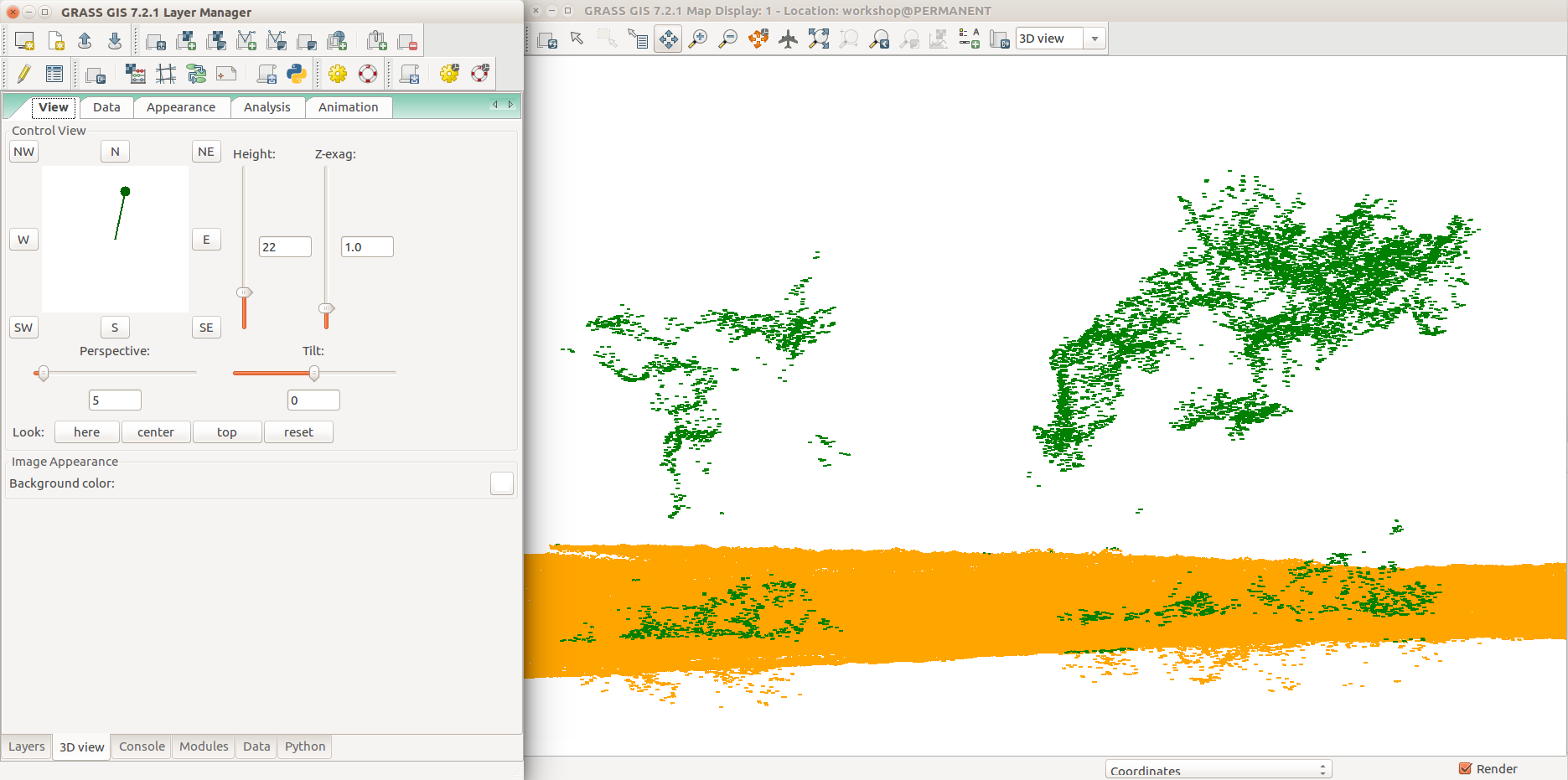
A menos que ya haya hecho esto, familiarícese con la vista 3D haciendo el ejercicio anterior. Luego importe todos los puntos pero esta vez no los añada al Administrador de capas (hay una caja de selección en la parte inferior del diálogo del módulo).
v.in.lidar -obt input=nc_tile_0793_016_spm.las output=points_all zrange=60,200
Ahora podemos explorar la nube de puntos en vista 3D:
- Deseleccione o remueva cualquier otra capa. (Cualquier capa en el Administrador de capas se intepreta como una superficie en la vista 3D.)
- Cambie a la vista 3D (en la esquina derecha del Visualizador de Mapas).
- Cuando 3D esté activo, Administrador de capas tiene un nuevo ícono para la configuración predeterminada del 3D, abrirla y cambiar.
- En la configuración, cambiar la forma de los puntos de esfera a X.
- Luego añadir vectorial points
- Puede necesitar ajutar el tamaño de los puntos si todos están sobrelapados.
- Ajustar la vista (perspectiva, altura).
- Ir de nuevo a la pestaña Vista y explorar las diferentes direcciones usando el control verde.
- Ir a la pestaña Apariencia y cambiar las condiciones de la luz (altura, dirección).
- Cuando termine, cambiar a la vista 2D.
-
Nube de puntos UAV clasificados por grupo de suelo (naranja) y no-suelo (verde).
-
Detalle mostrando árboles y puntos discrepantes.
Creación alternativa de DSM
Los primeros retornos no siempre representan la parte superior de la copa, pero pueden representar el primer golpe en algún lugar de la copa. Para tomar esto en cuenta creamos un ráster con el máximo de cada celda, en vez de importar los puntos en crudo:
r.in.lidar input=nc_tile_0793_016_spm.las output=maximum method=maximum return_filter=first
El ráster tiene muchas celdas faltantes, es por esto que queremos interpolar, pero al mismo tiempo, podemos reducir el número de puntos al reemplazar todos los puntos en una celda por solamente uno por celda. Podemos considerar la celda que represente un punto en el medio de la celda y usar r.to.vect para crear puntos a partir de estas celdas:
r.to.vect input=maximum output=points_for_dsm type=point -b
Ahora podemos decimar la nube de puntos, decimamos usando almacenado a ráster (binning). Interpolamos estos puntos para obtener el DSM:
v.surf.rst input=first_returns elevation=dsm tension=25 smooth=1 npmin=80
Explorar capas de vegetación en un ráster 3D
Definir la parte superior e inferior de la región computacional para que ajuste a la altura de la vegetación (estamos interesados en la vegetación etre 0 m y 30 m por encima del suelo).
g.region -p3 b=0 t=30
De manera similar al 2D, podemos realizar el almacenado (binning) también en tres dimensiones usando rásters 3D. Esto está implementado en un módulo llamado r3.in.lidar:
r3.in.lidar -d input=nc_tile_0793_016_spm.las n=count sum=intensity_sum mean=intensity proportional_n=prop_count proportional_sum=prop_intensity base_raster=terrain
Convertir las capas horizontales (rebanadas, planos) del ráster 3D en rásters 2D:
r3.to.rast input=prop_count output=slices_prop_count
Abrir un segundo Visualizador de mapas y añadir todos los rásters creados. Luego definir una tabla de colores consistente para todos.
r.colors slices_prop_count_001,slices_prop_count_002,... color=viridis
Ahora abra la Herramienta de Animación y añada todos los rásteres 2D ahí y use el deslizador (rebanador, slicer) para explorar las capas.
Optimizaciones, troubleshooting y limitaciones
Optimizaciones de velocidad:
- Rasterizar tempranamente.
- Para muchos usos, hay menos celdas que puntos. Adicionalmente, rásters pueden ser más rápidos, por ej. para visualizar, por que el ráster tiene un índice espacial natural. Finalmente, muchos algoritmos simplemente usan rásters.
- Si no puede rasterizar, vea si puede decimar (adelgazar, thin) la nube de puntos (usando v.in.lidar, r.in.lidar + r.to.vect, v.decimate)
- Almacenar (binning) (por ej. r.in.lidar, r3.in.lidar) es más rápido que interpolar y puede eliminar parte del análisis.
- La decimación rápida basda en conteo realiza casi siempre lo mismo en una nube de puntos dada para terreno en lugar de la decimación basada en malla (Petras et al. 2016).
- r.in.lidar
- Primero escoja la extensión y resolución de la región computacional
- tener suficiente memoria para evitar usar la opción percent (alto uso de memoria versus alto I/O)
- v.in.lidar
- -r limitar la importación a la extensión de la región
- -t no crear tabla de atributos
- -b no construir topología (aplicable a otros módulos también)
- -c guardar solamente las coordenadas, no categorías o IDs
Requisitos de memoria:
- para r.in.lidar
- depende del sentido de la salida y el tipo de análisis
- puede ser reducido por la opción percent
- ERROR: G_malloc: unable to allocate ... bytes of memory significa que no tiene suficiente memoria para la salida ráster, use la opción percent, una resolución más gruesa, o una región más pequeña
- en Linux la memoria disponible para procesos es RAM + SWAP (partición), pero cuando se usa SWAP el procesamiento es más lento
- para v.in.lidar
- menor cuando no construye la topología (bandera -b)
- no construir la topología mantiene bajo el uso de memoria, use export GRASS_VECTOR_LOWMEM=1 (os.environ['GRASS_VECTOR_LOWMEM'] = '1' en Python)
Límites:
- Elementos vectoriales con topología se limitan a 2 billones de elementos por mapa vectorial (2^31 - 1)
- Puntos sin topología se limitan solamente por el espacio del disco y lo que puedan procesar los módulos después. (Teóricamente, el conteo limitado solamente por la arquitectura 64bit que sería de 16 exbibytes por archivo, pero el valor depende de cada sistema de archivos.)
- Para vectoriales más grandes con atributos, se recomienda tomar PostgreSQL para los atributos (v.db.connect).
- No hay más límites para las versiones 32bit para operaciones que requiere memoria. (Desde 2016 hay una versión 64bit aún para MS Windows. Incluso la versión 32bit tiene una soporte de archivos grandes (LFS) y puede trabajar con archivos que excedan las limitaciones de 32bit.)
- Leer y escribir a disco (I/O) usualmente limita la velocidad. (Puede ser más rápido para rásters desde 7.2 usa diferentes algoritmos de compresión definidos con la variable GRASS_COMPRESSOR.
- Hay un límite para el número de archivos abiertos definido por el sistema operativo.
- El límite usualmente es 1024. En Linux se puede cambiar usando ulimit.
- Los módulos individuales pueden tener sus propias limitaciones o comportamientos con datos grandes basados en el algoritmo que usen. Sin embargo, en general los módulos están hechos para trabajar con grandes conjuntos de datos. Por ejemplo:
- r.watershed puede procesar 90,000 x 100,000 (9 billiones de celdas) en 77.2 hours (2.93GHz), y
- v.surf.rst puede procesar 1 million de puntos e interpolar 202,000 celdas en 13 minutes.
- Leer la documentación ya que provee descripciones de las limitaciones de cada módulo y cómo tratar con eso.
- Escriba a grass-user la lista de correos o a GRASS GIS bug tracker en caso de que llegue a algún límite. Por ejemplo, si tiene un número negativo como el número de putos o celdas, abra un ticket.
Bleeding edge

- Procesar nubes de puntos como rásters 3D
- r3.forestfrag
- r3.count.categories
- r3.scatterplot
- Perfil de puntos
- v.profile.points
- Integración PDAL
- prototipos: v.in.pdal, r.in.pdal
- meta: proveer acceso a algoritmos PDAL (filtros)
- r.in.kinect
- Tiene algunos elementos de r.in.lidar, v.in.lidar, and PCL (Point Cloud Library), pero la nube de puntos es continuamente actualizada desde el scanner Kinect usando OpenKinect libfreenect2 (usado en Tangible Landscape).
- Combinar datos de elevación de diferentes fuentes (ej. global y lidar DEM o lidar y UAV DEM)
- r.patch.smooth: Seamless fusion of high-resolution DEMs from two different sources
- r.mblend: Blending rasters of different spatial resolution
- mostrar grandes nubes de puntos en el Visualizador de mapas (2D)
- prototipo: d.points
- Guardar retorno e información de clase como categoría
- implementación experimental en v.in.lidar
- Decimación
- implementada: v.in.lidar, r.in.lidar
- v.decimate (con elementos experimentales)
Las contribuciones son más que bienvenidas. Puede discutirlas en la lista de correos grass-dev.
-
Tangible Landscape uses r.in.kinect to scan a sand model which is processed and analyzed in GRASS GIS and optionally visualized in Blender
-
Seamless fusion of high-resolution DEMs from multiple sources with r.patch.smooth: example of computing overlap zone for fusion
Ver también
- GRASS GIS at FOSS4G Boston 2017
- From GRASS GIS novice to power user (workshop at FOSS4G Boston 2017)
- Analytical data visualizations at ICC 2017
- Unleash the power of GRASS GIS at US-IALE 2017
- Creating animation from FUTURES output in GRASS GIS
- Introduction to GRASS GIS with terrain analysis examples
- Lidar Analysis of Vegetation Structure
- LIDAR
- GRASS GIS and Python
- GRASS GIS and R
- GRASS Location Wizard
Links externos:
- Processing lidar and general point cloud data in GRASS GIS (presentation slides)
- Seamless fusion of high-resolution DEMs from multiple sources (presentation slides)
- libLAS
- PDAL
Referencias:
- Brovelli M. A., Cannata M., Longoni U.M. 2004. LIDAR Data Filtering and DTM Interpolation Within GRASS. Transactions in GIS, April 2004, vol. 8, iss. 2, pp. 155-174(20), Blackwell Publishing Ltd. (full text at ResearchGate)
- Evans, J. S., Hudak, A. T. 2007. A Multiscale Curvature Algorithm for Classifying Discrete Return LiDAR in Forested Environments. IEEE Transactions on Geoscience and Remote Sensing 45(4): 1029 - 1038. (full text)
- Jasiewicz, J., Stepinski, T. 2013. Geomorphons - a pattern recognition approach to classification and mapping of landforms, Geomorphology. vol. 182, 147-156. DOI: 10.1016/j.geomorph.2012.11.005
- Leitão, J.P., Prodanovic, D., Maksimovic, C. 2016. Improving merge methods for grid-based digital elevation models. Computers & Geosciences, Volume 88, March 2016, Pages 115-131, ISSN 0098-3004. DOI: 10.1016/j.cageo.2016.01.001.
- Mitasova, H., Mitas, L. and Harmon, R.S., 2005, Simultaneous spline approximation and topographic analysis for lidar elevation data in open source GIS, IEEE GRSL 2 (4), 375- 379. (full text)
- Petras, V., Newcomb, D. J., Mitasova, H. 2017. Generalized 3D fragmentation index derived from lidar point clouds. In: Open Geospatial Data, Software and Standards. DOI: 10.1186/s40965-017-0021-8
- Petras, V., Petrasova, A., Jeziorska, J., Mitasova, H. 2016. Processing UAV and lidar point clouds in GRASS GIS. ISPRS - International Archives of the Photogrammetry, Remote Sensing and Spatial Information Sciences XLI-B7, 945–952, 2016 (full text at ResearchGate)
- Petrasova, A., Mitasova, H., Petras, V., Jeziorska, J. 2017. Fusion of high-resolution DEMs for water flow modeling. In: Open Geospatial Data, Software and Standards. DOI: 10.1186/s40965-017-0019-2
- Tabrizian, P., Harmon, B.A., Petrasova, A., Petras, V., Mitasova, H. Meentemeyer, R.K. Tangible Immersion for Ecological Design. In: ACADIA 2017: Proceedings of the 37th Annual Conference of the Association for Computer Aided Design in Architecture, 600-609. Cambridge, MA, 2017. (full text at ResearchGate)
- Tabrizian, P., Petrasova, A., Harmon, B., Petras, V., Mitasova, H., Meentemeyer, R. (2016, October). Immersive tangible geospatial modeling. In: Proceedings of the 24th ACM SIGSPATIAL International Conference on Advances in Geographic Information Systems (p. 88). ACM. DOI: 10.1145/2996913.2996950 (full text at ResearchGate)